ABOVE: modificeret fra © ISTOCK.com, tera vector
Næsten altid er det sværere at bygge noget op end at rive det ned. På samme måde er det en større udfordring at slå generne ind end at slå dem ud. Det er en realitet, som forskerne bliver nødt til at overvinde for at få mest muligt ud af genredigering. Ved at banke gener ind kan forskerne undersøge virkningerne af specifikke genvarianter, anvende reportergener som grønt fluorescerende protein til at spore genprodukter i tid og rum, undersøge genomets regulering og i sidste ende reparere sygdomsfremkaldende gener. “Det er en virkelig effektiv måde at undersøge hver eneste base i et gen på”, siger Greg Findlay, der er kandidat til en læge- eller ph.d.-grad ved University of Washington.
CRISPR-Cas9, en genredigeringsteknologi, der er kendt for sin brugervenlighed, kan slå generne ind eller ud. Ved at slå et gen ud, skal CRISPR-Cas9 indsættes i en celle ved hjælp af et guide-RNA, der målretter værktøjet mod det pågældende gen. Her skærer Cas9 genet over og klipper begge DNA-strenge over, hvorefter cellens normale DNA-reparationsmekanisme retter overskæringen ved hjælp af en proces kaldet non-homologous end joining (NHEJ). NHEJ er meget effektiv, men upræcis. Processen har en tendens til at indføre fejl i form af små indsættelser eller sletninger, som normalt er nok til at slå genet ud.
For at slå et gen ind, skal snittene imidlertid repareres meget præcist, uden ekstra indsættelser eller sletninger. Dette kræver, at man udnytter en anden DNA-reparationsmekanisme kaldet homologi-dirigeret reparation (HDR), som – i hvert fald i pattedyrceller – forekommer mindre effektivt, så dens hyppighed er mindre end NHEJ’s. Det komplicerer processen yderligere, at nogle genloci og celletyper i sagens natur er mindre gæstfrie for CRISPR-Cas9-redigering.
I de seneste par år har forskerne udviklet mange nye strategier til at øge effektiviteten af at banke i både store og små gener ved hjælp af CRISPR-Cas9, og undervejs har de foreslået og afprøvet nye anvendelsesmuligheder for denne type genredigering. Her udforsker The Scientist nogle af de mest lovende tilgange.
Vælg det
Forsker: Jon Chesnut, senior director of synthetic biology R&D, Thermo Fisher Scientific
Projekt: I forbindelse med udviklingen af et genmærkningssæt kaldet Truetag, som Thermo Fisher vil markedsføre senere i år, brugte Chesnut selekterbare markører til at forbedre
effektiviteten. En selekterbar markør – i dette tilfælde et antibiotikaresistensgen – sættes fast på et fluorescerende proteinmærke og indlægges i pattedyrceller. Disse celler dyrkes derefter i kultur med det tilhørende antibiotikum. Resistensgenet giver en selektiv fordel til de celler, der bærer det; kun de er i stand til at vokse, og derfor indeholder de celler, der vokser, det pågældende genmærke. Selv hvis effektiviteten af genindsættelsen er lav, kan forskerne bruge antibiotikaudvælgelse i en uge eller mere for at ende med en høj procentdel af celler med vellykkede indsættelser.
Ved at bruge antibiotikaet puromycin eller blasticidin sammen med sættet lykkedes det Chesnuts hold at øge genindsættelsesraten fra 10-30 procent til 90 procent eller mere i nogle cellepopulationer. Et par særligt vanskelige gener gik fra en indsættelsesrate på mindre end 1 procent til mere end 90 procent. Det er vigtigt at teste flere doser antibiotika på den cellelinje, man planlægger at bruge, for at finde den rigtige dosis, siger Chesnut: man ønsker at dræbe celler uden indsættelser, men ikke celler med vellykkede indsættelser.
Try It: Selekterbare markører fungerer bedst, når genet af interesse er stærkt udtrykt, siger Chesnut. “Hvis det ikke er det, kan du stadig få selektion, men du får måske ikke nok ekspression af dit fluorescerende proteinmærke til at kunne detektere det.” De generelle begrænsninger for CRISPR-Cas9 gælder også. “Der er regioner af genomet, der ikke skærer særlig godt med CRISPR, og vi er stadig ikke sikre på hvorfor,” tilføjer han. Og nogle celletyper accepterer ikke let fremmed DNA, RNA eller RNA-proteinkomplekser – de tre metoder til levering af CRISPR-Cas9.
For at få mere held med at indsætte selekterbare markører skal man sørge for, at der er en såkaldt PAM-sekvens, et kort tag i mål-DNA’et, som CRISPR-Cas9 skal genkende, før det skærer, inden for 10 basepar af det ønskede genindsættelsessted, siger Chesnut. Hvis man er længere væk fra klipningsstedet end det, kan effektiviteten af indsættelsen være for lav til at være funktionel. Uden et PAM-sted kan man prøve TALENs eller zinkfingernukleaser, selv om disse ældre genredigeringsteknikker er mere vanskelige end CRISPR.
Timed Inhibition
Forsker: Jacob Corn, genombiolog, Swiss Federal Institute of Technology, Zürich
Projekt: Forskere forstår ikke, hvorfor NHEJ-vejen i høj grad udkonkurrerer HDR-vejen i pattedyrceller. “Gær laver HDR som en gal”, siger Corn. I et forsøg på at sætte gang i denne DNA-reparationsproces i menneskelige celler og forbedre kontrollen med gen-knock-in forsøger han og hans team at finde ud af, hvordan HDR reguleres. De screenede menneskelige celler for gener, hvis knockdown førte til øget HDR i cellen, og søgte derefter efter små molekylære inhibitorer af disse gener. Et af de gener, der dukkede op, koder for CDC7, en kinase, der regulerer cellecyklusens overgang til S-fasen; dets inhibitor, XL413, øgede genets knock-in effektivitet to- eller tredobbelt (BioRXiv, DOI: 10.1101/500462, 2018). Det skyldes, at HDR kun forekommer i nogle dele af cellecyklusen, herunder S-fasen, siger Corn. Hvis du tilføjer inhibitoren XL413 samtidig med, at du bruger CRISPR-Cas9 til at redigere dit målgen, hober cellerne sig op i fasen umiddelbart før S-fasen. Hvis man så fjerner XL413, går alle cellerne ind i S-fasen og øger knock-in-effektiviteten.

Corn har anvendt denne teknik i mange udødelige menneskelige cellelinjer og i menneskelige T-celler. Den kan banke i korte DNA-strækninger, såsom SNP’er, såvel som i store gener. Der er ingen grund til, at det ikke skulle virke i mus, siger han, selv om han ikke har afprøvet det.
Try It: “Timing er helt afgørende,” siger Corn. Cas9 skal klippe DNA’et samtidig med, at XL413 tilsættes. Hvis du hæmmer først og derefter frigiver, mens du redigerer med CRISPR-Cas9, falder homolog rekombinationseffektiviteten tre gange i stedet for at stige, fordi cellerne frigives i den forkerte fase af cellecyklussen.
Og som med enhver HDR-indsats, siger Corn, skal du altid køre en no-nuclease-kontrol for at sikre, at du ikke ved et uheld forstærker kontaminerende DNA, der flyder rundt i dit laboratorium. Når du har introduceret knock-in’en, skal du “sekventere, sekventere, sekventere, sekventere, sekventere,” siger han. Det kan give bagslag at bruge et reporter-system som f.eks. et fluorescerende proteintag til at demonstrere en vellykket genindsættelse. Sekventering verificerer, at indsættelserne blev foretaget på det rigtige sted.
Spiller det lange spil
Forsker: Forsker: Channabasavaiah Gurumurthy, direktør for musens genome engineering core facility, University of Nebraska Medical Center
Projekt: For et par år siden fik Gurumurthy og hans kolleger en åbenbaring, da de overvejede, hvor vanskeligt det var at indbygge gener i zygoter af mus, mens de forsøgte at gøre det.
Forskerne havde succes med at indsætte kort, enkeltstrenget DNA, så hvorfor ikke prøve at lave en knock-in ved at indsætte langt, enkeltstrenget DNA? Faktisk øger tilgangen, som Gurumurthy kalder Easi-CRISPR (efficient additions with ssDNA inserts -CRISPR), effektiviteten med 2,5 gange, og brugen af enkeltstrenget DNA skærer hastigheden af off-target-indsættelser 100-foldigt i cellekultur (Nat Protoc 13:195-215, 2018; Nature 559:405-09, 2018). “Det er ret enormt,” siger han. I Gurumurthys laboratorium har Easi-CRISPR genereret en knock-in muselinje for 9 ud af 10 gener, som de har forsøgt. En samarbejdspartner har også brugt det i menneskelige T-celler til at skabe CAR-T-celler, patientspecifikke immunceller til bekæmpelse af kræft.
Try It: Easi-CRISPR er langt fra idiotsikker, advarer Gurumurthy. Nogle gange indsætter teknikken kun en del af genet. Han tilføjer, at den også kan forvrænge homologiarmene – de korte sekvenser på hver side af genet, som fører det hjem til dets korrekte mål i genomet. Og nogle loci er uforklarligt nok vanskeligere at indsætte end andre.
Få kommercielle leverandører designer og syntetiserer brugerdefinerede lange, enkeltstrengede DNA-strenge. Du kan lave dit eget, men stabiliteten af enkeltstrenget DNA varierer; mindre stabile sekvenser vil have lavere udbytte, så du skal muligvis syntetisere flere af dem, siger Gurumurthy.
Forskere, der ikke er i stand til at indsætte CRISPR i enkeltcellede museembryoner, kan betale en kernefacilitet for at lave musene med deres DNA-sekvens, siger Gurumurthy. Kernefaciliteter som hans tager mellem 5.000 og 15.000 dollars for at generere et eller to avlspar; kommercielle faciliteter tager 20.000 og 50.000 dollars, siger han.
Knock-in By Numbers
Forsker: Greg Findlay, MD/PhD-kandidat i Jay Shendures laboratorium, University of Washington
Projekt: Findlay og hans kolleger havde til formål at forbedre den måde, hvorpå klinikere fortolker mutationer i bryst- og æggestokkræftgenet BRCA1. Dette gen har tusindvis af varianter, men forskerne ved ikke, hvordan de fleste af dem påvirker dets funktion. For at undersøge virkningen af disse varianter brugte de en knock-in-teknik, som de udviklede, kaldet saturation genome editing (Nature, 562:217-22, 2018).
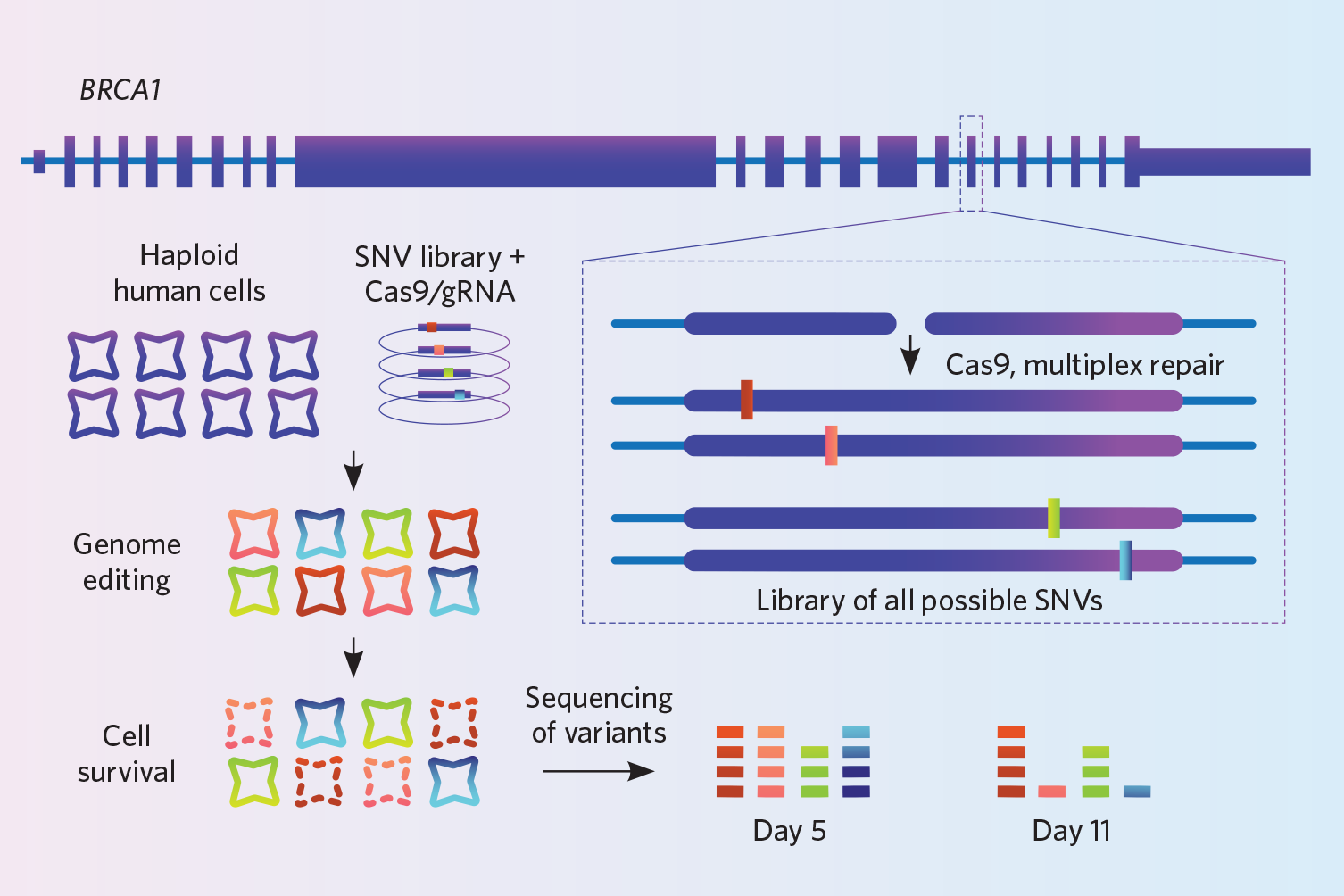
I en udødeliggjort haploid menneskelig cellelinje brugte de CRISPR-Cas9 til at banke 4.000 små varianter ind i millioner af celler på en gang in vitro. Genomet bliver skåret det samme sted i hver celle, men hver celle får en anden variant i sit genom. For at fremme HDR slog de også ligase4-genet ud og deaktiverede dermed NHEJ-reparationsvejen – et trin, der gav en tredobbelt stigning i effektiviteten, siger Findlay. Da alle cellernes knock-ins er forskellige, sekventerede de til sidst cellerne grundigt og dækkede den samme genomiske region millioner af gange for at sikre sig, at de faktisk havde slået de 4.000 varianter ind, som de ønskede at undersøge. De sekventerede på to tidspunkter og udledte, at de knock-ins, der ikke dukkede op i sekventeringen på det andet tidspunkt, var dem, der forstyrrede genets funktion, fordi de celler, der bar dem, måtte være døde.
Try It: Findlays hold fik fremstillet DNA-oligos for de 4.000 varianter til dem på et mikroarray. Du kan købe arrays med 6.000 til 250.000 oligoer, så overvej at få mere for pengene ved at kombinere flere eksperimenter på det samme array, siger Findlay. Deres laboratorium betaler omkring 5.000 dollars for 100.000 oligoer.
Denne strategi er forbundet med begrænsninger: Den er indtil videre kun blevet brugt til at banke enkelt-nucleotidvarianter ind, og alle ændringerne skal være i det samme gen. Metoden fungerer bedst, når man redigerer et ret smalt område af DNA, ca. 110-120 basepar, fordi længere DNA-oligoer ville have for mange fejl, siger Findlay. Det er også vigtigt at sekventere meget dybt for at sikre, at man tager højde for det fulde antal varianter, som man havde til hensigt at banke ind.