ABOVE: modified from © ISTOCK.com, tera vector
Nästan alltid är det svårare att bygga något än att riva ner det. På samma sätt är det en större utmaning att slå in gener än att slå ut dem. Det är en verklighet som forskarna måste övervinna för att få ut så mycket som möjligt av genredigering. Genom att slå in gener kan forskarna studera effekterna av specifika genvarianter, använda reportergener som grönt fluorescerande protein för att spåra genprodukter i tid och rum, undersöka genomets reglering och slutligen reparera sjukdomsframkallande gener. ”Det är ett mycket effektivt sätt att undersöka varje bas i en gen”, säger Greg Findlay, doktorand vid University of Washington.
CRISPR-Cas9, en genredigeringsteknik som är känd för sin användarvänlighet, kan slå in eller ut gener. Att slå ut en gen innebär att CRISPR-Cas9 förs in i en cell med hjälp av ett guide-RNA som riktar verktyget mot den aktuella genen. Där klipper Cas9 genen genom båda DNA-strängarna, och cellens normala DNA-reparationsmekanism reparerar genen med hjälp av en process som kallas NHEJ (non-homologous end joining). NHEJ är mycket effektiv men felaktig. Processen tenderar att införa fel i form av små insättningar eller deletioner som vanligtvis är tillräckliga för att slå ut genen.
För att slå in en gen måste dock snitten repareras mycket exakt, utan extra insättningar eller deletioner. Detta kräver att man utnyttjar en andra DNA-reparationsmekanism som kallas homologiskt riktad reparation (HDR), som åtminstone i däggdjursceller förekommer mindre effektivt, så dess frekvens är dvärglik den för NHEJ. Det faktum att vissa genloci och celltyper är mindre mottagliga för CRISPR-Cas9-redigering komplicerar processen ytterligare.
Under de senaste åren har forskarna utvecklat många nya strategier för att öka effektiviteten när det gäller att slå in både stora och små gener med hjälp av CRISPR-Cas9, och på vägen dit har de föreslagit och testat nya tillämpningar för denna typ av genredigering. Här undersöker The Scientist några av de mest lovande metoderna.
Välj det
Forskare: Jon Chesnut, senior director of synthetic biology R&D, Thermo Fisher Scientific
Projekt: Chesnut använde selekterbara markörer för att förbättra
effektiviteten när han utvecklade ett genmärkningskit kallat Truetag som Thermo Fisher kommer att släppa ut på marknaden senare i år. En selekterbar markör – i det här fallet en gen för antibiotikaresistens – fästs på en fluorescerande proteinmärkning och slås in i däggdjursceller. Dessa celler odlas sedan i kultur med det tillhörande antibiotikumet. Resistensgenen ger en selektiv fördel till de celler som bär den; de är de enda som kan växa, och därför innehåller de som växer den intressanta genmärkningen. Även om effektiviteten av geninsättning är låg kan forskarna använda antibiotikaurval i en vecka eller mer för att i slutändan få en hög andel celler med lyckade insättningar.
Om man använde antibiotikan puromycin eller blasticidin tillsammans med satsen lyckades Chesnuts team öka geninsättningstakten från 10-30 procent till 90 procent eller mer i vissa cellpopulationer. Några särskilt svåra gener gick från en insättningsgrad på mindre än 1 procent till mer än 90 procent. Det är viktigt att testa flera doser av antibiotika på den cellinje du planerar att använda för att hitta rätt dos, säger Chesnut: du vill döda celler utan insättningar men inte celler med lyckade insättningar.
Try It: Selekterbara markörer fungerar bäst när genen av intresse är starkt uttryckt, säger Chesnut. ”Om den inte är det kan du fortfarande få selektion, men du kanske inte får tillräckligt mycket uttryck av din fluorescerande proteinmarkering för att kunna detektera den.” Dessutom gäller de allmänna begränsningarna för CRISPR-Cas9. ”Det finns regioner av genomet som inte skärs så bra med CRISPR, och vi vet fortfarande inte varför”, tillägger han. Och vissa celltyper accepterar inte lätt främmande DNA, RNA eller RNA-proteinkomplex – de tre metoderna för CRISPR-Cas9-leverans.
För att få mer tur med att infoga selekterbara markörer ska du se till att det finns en så kallad PAM-sekvens, en kort tagg i mål-DNA:t som CRISPR-Cas9 måste känna igen innan den skär, inom 10 baspar av den önskade geninförselplatsen, säger Chesnut. Om man befinner sig längre bort än så från skärningsplatsen kan insättningseffektiviteten vara för låg för att vara funktionell. Utan en PAM-plats kan man prova TALENs eller zinkfingernukleaser, även om dessa äldre genredigeringstekniker är knepigare än CRISPR.
Timed Inhibition
Forskare: Jacob Corn, genombiolog, Swiss Federal Institute of Technology, Zürich
Projekt: ”Hämning av en cell i en cell: Forskare förstår inte varför NHEJ-vägen konkurrerar ut HDR-vägen i däggdjursceller. ”Jäst gör HDR som en galning”, säger Corn. I ett försök att få fart på denna DNA-reparationsprocess i mänskliga celler och förbättra kontrollen av genernas knock-in-teknik försöker han och hans grupp fastställa hur HDR regleras. De undersökte mänskliga celler efter gener vars knockdown ledde till ökad HDR i cellen och sökte sedan efter små molekylära hämmare av dessa gener. En av de gener som dök upp kodar för CDC7, ett kinas som reglerar cellcykelns övergång till S-fasen; dess hämmare, XL413, ökade genernas knock-in-effektivitet två- eller tredubbelt (BioRXiv, DOI: 10.1101/500462, 2018). Det beror på att HDR endast förekommer i vissa delar av cellcykeln, inklusive S-fasen, säger Corn. Om du lägger till inhibitorn XL413 samtidigt som du använder CRISPR-Cas9 för att redigera din målgen, samlas cellerna i fasen omedelbart före S-fasen. Om du sedan tar bort XL413 går alla celler in i S-fasen och ökar knock-in-effektiviteten.

Corn har använt denna teknik i många odödliga humana cellinjer och i humana T-celler. Den kan knacka in korta DNA-sträckor, t.ex. SNPs, såväl som stora gener. Det finns ingen anledning till att det inte skulle fungera på möss, säger han, även om han inte har testat det.
Try It: ”Timing är helt avgörande”, säger Corn. Cas9 måste klippa DNA:t samtidigt som XL413 tillsätts. Om du hämmar först och sedan släpper ut medan du redigerar med CRISPR-Cas9, sjunker den homologa rekombinationseffektiviteten tre gånger i stället för att öka, eftersom cellerna släpps ut i fel fas av cellcykeln.
Och som med alla HDR-försök säger Corn att du alltid ska köra en kontroll utan nukleas för att se till att du inte oavsiktligt amplifierar kontaminerande DNA som flyter runt i ditt labb. Efter att du har introducerat knock-in-produkten ska du ”sekvensera, sekvensera, sekvensera, sekvensera, sekvensera”, säger han. Att bara använda ett rapporterande system, t.ex. en fluorescerande proteintagg, för att visa att man lyckats föra in genen kan ge bakslag. Sekvensering verifierar att insatserna gjordes på rätt plats.
Playing the Long Game
Forskare: Channabasavaiah Gurumurthy, chef för musens genome engineering core facility, University of Nebraska Medical Center
Projekt: ”Den långa vägen”: För några år sedan, när Gurumurthy och hans kollegor funderade över svårigheten att slå in gener och samtidigt försöka göra det i musens zygoter, fick Gurumurthy och hans kollegor en uppenbarelse.
Forskarna lyckades föra in kort, enkelsträngat DNA, så varför inte försöka göra en knock-in genom att föra in långt, enkelsträngat DNA? Metoden, som Gurumurthy kallar Easi-CRISPR (efficient additions with ssDNA inserts -CRISPR), ökar effektiviteten med 2,5 gånger, och genom att använda enkelsträngat DNA minskar antalet insättningar utanför måltavlan 100 gånger i cellkultur (Nat Protoc 13:195-215, 2018; Nature 559:405-09, 2018). ”Det är ganska enormt”, säger han. I Gurumurthys labb har Easi-CRISPR genererat en knock-in-muslinje för 9 av 10 gener som de har provat. En medarbetare har också använt det i mänskliga T-celler för att skapa CAR-T-celler, patientspecifika immunceller för att bekämpa cancer.
Try It: Easi-CRISPR är långt ifrån idiotsäkert, varnar Gurumurthy. Ibland infogar tekniken endast en del av genen. Dessutom, tillägger han, kan den förvränga homologiarmarna – de korta sekvenser på vardera sidan av genen som leder den till rätt mål i genomet. Och vissa loci är oförklarligt nog svårare att infoga än andra.
Få kommersiella leverantörer utformar och syntetiserar skräddarsytt långt, enkelsträngat DNA. Du kan göra ditt eget, men stabiliteten hos enkelsträngat DNA varierar; mindre stabila sekvenser kommer att ge lägre avkastning, så du kan behöva syntetisera fler av dem, säger Gurumurthy.
Forskare som inte kan infoga CRISPR i encelliga musembryon kan betala en kärnanläggning för att göra möss med deras DNA-sekvens, säger Gurumurthy. En kärnanläggning som hans tar mellan 5 000 och 15 000 dollar för att skapa ett eller två avelspar, medan kommersiella anläggningar tar mellan 20 000 och 50 000 dollar, säger han.
Knock-in By Numbers
Forskare: Greg Findlay, doktorand i medicin i Jay Shendures labb, University of Washington
Projekt: Projekt: Findlay och hans kollegor ville förbättra hur kliniker tolkar mutationer i bröst- och äggstockscancergenen BRCA1. Den genen har tusentals varianter, men forskarna vet inte hur de flesta av dem påverkar dess funktion. För att studera effekten av dessa varianter använde de en knock-in-teknik som de utvecklat och som kallas saturation genome editing (Nature, 562:217-22, 2018).
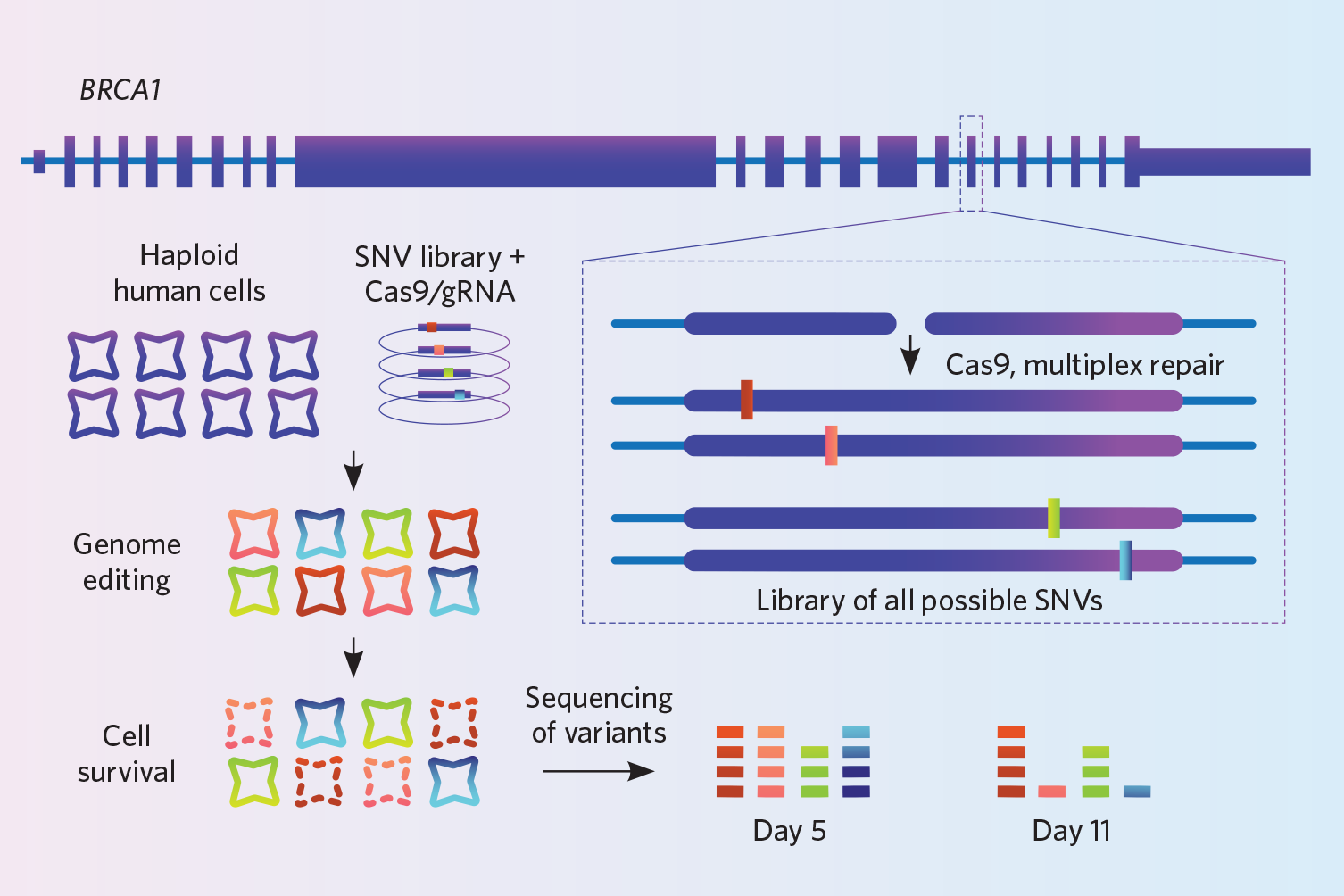
I en odödlig haploid mänsklig cellinje använde de CRISPR-Cas9 för att knacka in 4 000 små varianter i miljontals celler på en gång in vitro. Genomet skärs av på samma ställe i varje cell, men varje cells genom får en annan variant. För att främja HDR slog de också ut genen ligas4, vilket inaktiverade NHEJ-reparationsvägen – ett steg som gav en trefaldig ökning av effektiviteten, säger Findlay. Slutligen, eftersom alla cellernas knock-ins är olika, sekvenserade de cellerna djupt, genom att täcka samma genomiska region miljontals gånger, för att försäkra sig om att de verkligen slog in de 4 000 varianter som de ville studera. De sekvenserade vid två tidpunkter och drog slutsatsen att de knock-ins som inte dök upp i sekvenseringen vid den andra tidpunkten var sådana som störde genens funktion, eftersom de celler som bar på dem måste ha dött.
Prova det: Findlays grupp lät tillverka DNA-oligos för de 4 000 varianterna för dem på ett mikroarray. Du kan köpa matriser med 6 000 till 250 000 oligos, så överväg att få mer för pengarna genom att kombinera flera experiment på samma matris, säger Findlay. Deras labb betalar cirka 5 000 dollar för 100 000 oligos.
Denna strategi har begränsningar: den har hittills bara använts för att slå in varianter av enstaka nukleotider, och alla ändringar måste vara i samma gen. Metoden fungerar bäst när man redigerar en ganska smal region av DNA, cirka 110-120 baspar, eftersom längre DNA-oligos skulle ha för många fel, säger Findlay. Det är också viktigt att sekvensera mycket djupt för att se till att du tar hänsyn till hela antalet varianter som du hade för avsikt att knacka in.