ABOVE: modified from © ISTOCK.com, tera vector
Majdnem mindig nehezebb felépíteni valamit, mint lebontani. Hasonlóképpen a gének kiütése nagyobb kihívást jelent, mint a kiütésük. Ezt a valóságot a kutatóknak le kell győzniük ahhoz, hogy a legtöbbet hozzák ki a génszerkesztésből. A gének bekopogtatása lehetővé teszi a tudósok számára, hogy tanulmányozzák az egyes génváltozatok hatásait, hogy riportergénekkel, például zöld fluoreszcens fehérjével időben és térben nyomon kövessék a géntermékeket, hogy megvizsgálják a genom szabályozását, és végül, hogy megjavítsák a betegséget okozó géneket. “Ez egy igazán hatékony módja annak, hogy egy gén minden bázisát kikérdezzük” – mondja Greg Findlay, a Washingtoni Egyetem doktorjelöltje.
A felhasználóbarátságáról ismert génszerkesztő technológia, a CRISPR-Cas9 képes gének ki- vagy bekapcsolására. Egy gén kiütése azt jelenti, hogy a CRISPR-Cas9-et beillesztik egy sejtbe egy vezető RNS segítségével, amely az eszközt a kívánt génre irányítja. Ott a Cas9 elvágja a gént, átvágva a DNS mindkét szálát, és a sejt szokásos DNS-javító mechanizmusa a nem-homológ végcsatlakozásnak (NHEJ) nevezett folyamat segítségével kijavítja a vágást. Az NHEJ rendkívül hatékony, de pontatlan. A folyamat hajlamos hibákat bevinni kis beillesztések vagy deléciók formájában, amelyek általában elegendőek a gén kiütéséhez.
A gén kiütéséhez azonban a vágásokat nagyon pontosan kell kijavítani, extra beillesztések vagy deléciók nélkül. Ehhez egy második DNS-javító mechanizmus, a homológia-irányított javítás (HDR) kihasználására van szükség, amely – legalábbis az emlősök sejtjeiben – kevésbé hatékonyan működik, így gyakorisága eltörpül az NHEJ gyakorisága mellett. Tovább bonyolítja a folyamatot az a tény, hogy egyes génlókuszok és sejttípusok eleve kevésbé alkalmasak a CRISPR-Cas9 szerkesztésre.
Az elmúlt néhány évben a kutatók számos új stratégiát dolgoztak ki a kisebb és nagyobb gének CRISPR-Cas9 segítségével történő kiütésének hatékonyságának növelésére, és eközben új alkalmazásokat javasoltak és teszteltek az ilyen típusú génszerkesztésre. A The Scientist itt a legígéretesebb megközelítések közül vizsgál meg néhányat.
Válassza ki
Kutató: Jon Chesnut, a Thermo Fisher Scientific szintetikus biológia K&D vezető igazgatója
Projekt: Chesnut a Truetag nevű génjelölő készlet kifejlesztése során, amelyet a Thermo Fisher még ebben az évben forgalomba hoz, szelektálható markereket használt a hatékonyság
javítására. Egy szelektálható markert – ebben az esetben egy antibiotikum-rezisztencia gént – egy fluoreszcens fehérje címkéhez ragasztanak és beütik
emlőssejtekbe. Ezeket a sejteket ezután a hozzájuk tartozó antibiotikummal együtt tenyésztik. A rezisztenciagén szelektív előnyt biztosít az azt hordozó sejteknek; csak ezek képesek növekedni, és így azok, amelyek növekednek, tartalmazzák a kívánt géncímkét. Még ha a génbeillesztés hatékonysága alacsony is, a kutatók egy hétig vagy még tovább is használhatják az antibiotikumos szelekciót, hogy végül a sikeres beillesztést végző sejtek nagy százalékát kapják.
A puromycin vagy blasticidin antibiotikumot a készlettel együtt használva Chesnut csapatának sikerült a génbeillesztés arányát 10-30 százalékról 90 százalékra vagy még többre növelnie egyes sejtpopulációkban. Néhány különösen nehéz gén esetében a beillesztési arány az 1 százalék alatti értékről 90 százalék fölé emelkedett. Chesnut szerint fontos, hogy több antibiotikum-dózist teszteljünk a használni kívánt sejtvonalon, hogy megtaláljuk a megfelelő dózist: a beillesztés nélküli sejteket el akarjuk pusztítani, de a sikeres beillesztéssel rendelkező sejteket nem.
Kipróbáljuk: Chesnut szerint a szelektálható markerek akkor működnek a legjobban, ha a kívánt gén erősen kifejeződik. “Ha nem így van, akkor is szelekciót kaphatsz, de lehet, hogy a fluoreszcens fehérjemaradványod nem expresszálódik eléggé ahhoz, hogy detektálni tudd”. A CRISPR-Cas9 általános korlátai is érvényesek. “A genomnak vannak olyan régiói, amelyeket a CRISPR nem vág túl jól, és még mindig nem tudjuk, hogy miért” – teszi hozzá. És egyes sejttípusok nem fogadják el könnyen az idegen DNS-t, RNS-t vagy RNS-fehérje komplexeket – a CRISPR-Cas9 szállításának három módszerét.
A szelektálható markerek szerencsésebb beillesztéséhez győződjön meg arról, hogy a cél-DNS-ben van egy úgynevezett PAM-szekvencia, egy rövid tag, amelyet a CRISPR-Cas9-nek fel kell ismernie, mielőtt vág, 10 bázispáron belül a kívánt gén beillesztési helyétől, mondja Chesnut. Ennél távolabb a vágási helytől, és a beillesztés hatékonysága túl alacsony lehet ahhoz, hogy működőképes legyen. PAM-hely nélkül megpróbálkozhatunk a TALEN-ekkel vagy a cinkujjas nukleázokkal, bár ezek a régebbi génszerkesztési technikák trükkösebbek, mint a CRISPR.
Timed Inhibition
Kutató: Jacob Corn, genombiológus, Svájci Szövetségi Technológiai Intézet, Zürich
Projekt: A kutatók nem értik, hogy az NHEJ-útvonal miért versenyez nagymértékben a HDR-útvonallal az emlőssejtekben. “Az élesztők őrült módon végzik a HDR-t” – mondja Corn. Annak érdekében, hogy felpörgessék ezt a DNS-javítási folyamatot az emberi sejtekben, és javítsák a génbecsapódás ellenőrzését, ő és csapata megpróbálja pontosan meghatározni, hogyan szabályozódik a HDR. Olyan géneket kerestek emberi sejtekben, amelyek kiütése a sejtben megnövekedett HDR-hez vezetett, majd kis molekulájú gátlókat kerestek ezeknek a géneknek. Az egyik felbukkant gén a CDC7-et kódolja, egy olyan kinázt, amely a sejtciklus S-fázisba való átmenetét szabályozza; gátlója, az XL413 két-háromszorosára növelte a gén kopogtatásának hatékonyságát (BioRXiv, DOI: 10.1101/500462, 2018). Ez azért van így, mert a HDR a sejtciklusnak csak bizonyos részein, köztük az S-fázisban fordul elő, mondja Corn. Ha az XL413 gátlót akkor adjuk hozzá, amikor a CRISPR-Cas9 segítségével szerkesztjük a célgént, a sejtek az S-fázist közvetlenül megelőző fázisban halmozódnak fel. Ezután eltávolítjuk az XL413-at, és az összes sejt az S-fázisba kerül, ami növeli a kopogtatás hatékonyságát.

Corn ezt a technikát számos immortalizált emberi sejtvonalon és emberi T-sejteken alkalmazta. Rövid DNS-szakaszok, például SNP-k, valamint nagyméretű gének kopogtatására is képes. Nincs ok arra, hogy ne működjön egereken is, mondja, bár még nem tesztelte.
Kipróbáljuk: “Az időzítés abszolút kulcsfontosságú” – mondja Corn. A Cas9-nek ugyanabban az időben kell elvágnia a DNS-t, amikor az XL413-at hozzáadjuk. Ha a CRISPR-Cas9-szel történő szerkesztés során először gátoljuk, majd felszabadítjuk, a homológ rekombináció hatékonysága ahelyett, hogy növekedne, háromszorosára csökken, mivel a sejtek a sejtciklus rossz fázisába kerülnek.
És mint minden HDR-erőfeszítésnél, Corn szerint mindig futtassunk egy nukleázmentes kontrollt, hogy megbizonyosodjunk arról, hogy véletlenül sem erősítjük fel a laboratóriumban keringő szennyező DNS-t. A knock-in bevezetése után “szekvencia, szekvencia, szekvencia, szekvencia” – mondja. Egy riporterrendszer, például egy fluoreszcens fehérjecímke használata a sikeres génbeillesztés demonstrálására visszafelé sülhet el. A szekvenálás igazolja, hogy a beillesztés a megfelelő helyen történt.
A hosszú játék
Kutató: Channabasavaiah Gurumurthy, a University of Nebraska Medical Center
Projekt: Gurumurthy és munkatársai néhány évvel ezelőtt, miközben azon töprengtek, hogy milyen nehéz a gének bekopogtatása, miközben ezt egér zigótákba próbálták beültetni, felfedezést tettek.
A kutatók sikeresen beillesztettek rövid, egyszálú DNS-t, miért ne próbálhatnának hát hosszú, egyszálú DNS beillesztésével kopogtatást végezni? Valóban, a megközelítés, amelyet Gurumurthy Easi-CRISPR-nek (efficient additions with ssDNA inserts -CRISPR) nevez, 2,5-szeresére növeli a hatékonyságot, és az egyszálú DNS használata 100-szorosára csökkenti a célponton kívüli inszerciók arányát sejtkultúrában (Nat Protoc 13:195-215, 2018; Nature 559:405-09, 2018). “Ez egészen hatalmas” – mondja. Gurumurthy laboratóriumában az Easi-CRISPR az általuk kipróbált 10 génből 9-re létrehozta a knock-in egérvonalat. Egy munkatársa emberi T-sejtekben is alkalmazta, hogy CAR-T sejteket hozzon létre, a rák elleni küzdelemre alkalmas, páciensspecifikus immunsejteket.
Kipróbálja: Az Easi-CRISPR messze nem bolondbiztos, figyelmeztet Gurumurthy. Előfordul, hogy a technika csak a gén egy részét ülteti be. Azt is hozzáteszi, hogy megzavarhatja a homológia karokat – a gén két oldalán lévő rövid szekvenciákat, amelyek a megfelelő célponthoz vezetik a gént a genomban. És egyes lokuszokat megmagyarázhatatlanul nehezebb beilleszteni, mint másokat.
Néhány kereskedelmi forgalmazó tervez és szintetizál egyedi hosszú, egyszálú DNS-t. Lehet sajátot készíteni, de az egyszálú DNS stabilitása változó; a kevésbé stabil szekvenciáknak kisebb lesz a hozama, ezért lehet, hogy többet kell szintetizálni belőlük, mondja Gurumurthy.
A kutatók, akik nem tudják a CRISPR-t beilleszteni egysejtű egérembriókba, fizethetnek egy központi létesítménynek, hogy készítsen egereket az ő DNS-szekvenciájukkal, mondja Gurumurthy. Az olyan alaplétesítmények, mint az övé, 5000-15 000 dollárt kérnek egy vagy két tenyészpár létrehozásáért; a kereskedelmi létesítmények 20 000-50 000 dollárt kérnek, mondja.
Knock-in By Numbers
Kutató: Greg Findlay, MD/PhD jelölt Jay Shendure laboratóriumában, Washington Egyetem
Projekt: Findlay és kollégái célja az volt, hogy javítsák, hogyan értelmezik a klinikusok az emlő- és petefészekrák BRCA1 génjének mutációit. Ennek a génnek több ezer változata van, de a kutatók nem tudják, hogy ezek többsége hogyan befolyásolja a működését. E variánsok hatásának tanulmányozására az általuk kifejlesztett, telítési genomszerkesztésnek nevezett knock-in technikát alkalmazták (Nature, 562:217-22, 2018).
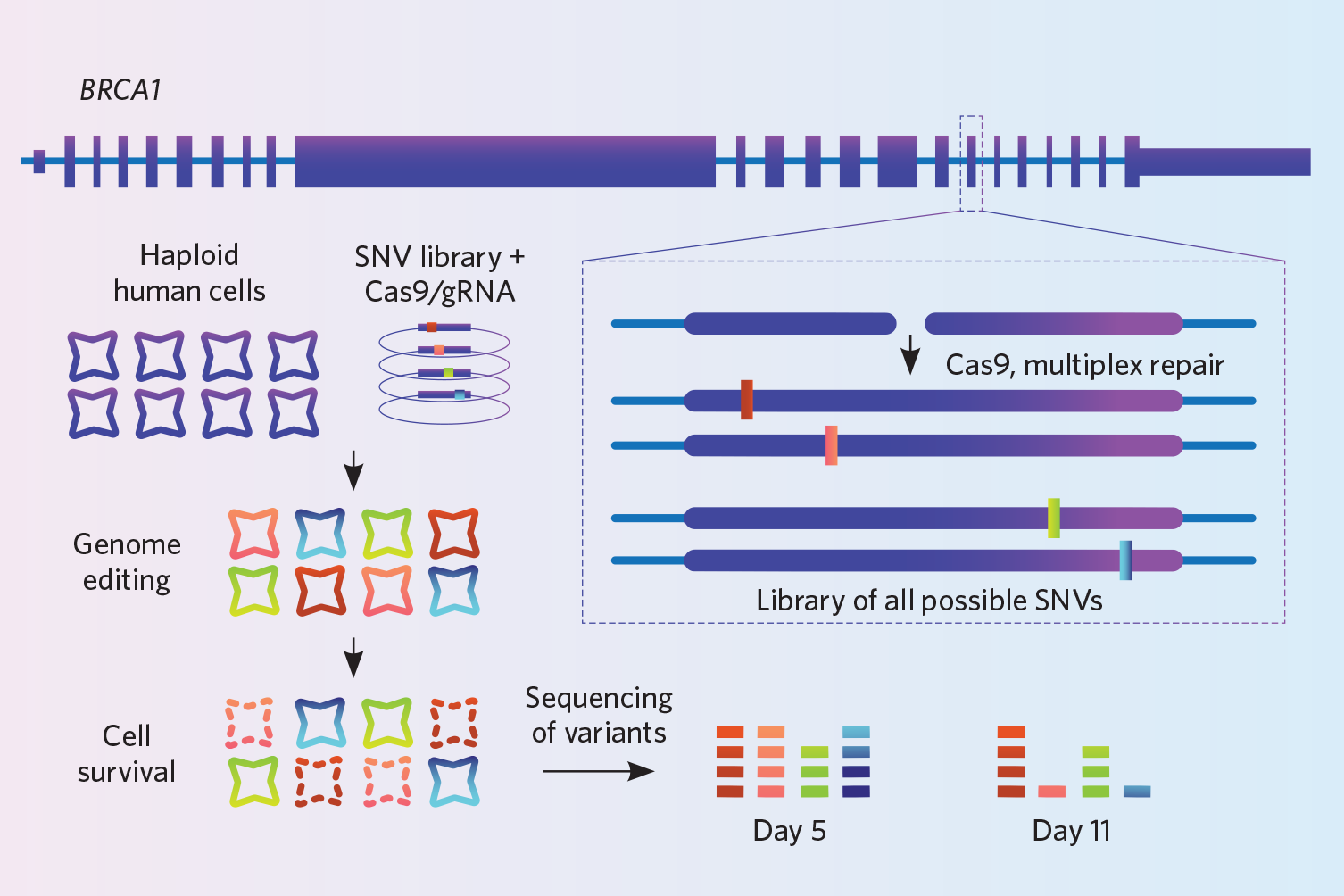
Egy immortalizált haploid emberi sejtvonalban a CRISPR-Cas9 segítségével egyszerre 4 000 apró variánst ütöttek be több millió sejtben in vitro. A genomot minden sejtben ugyanazon a helyen vágták el, de minden sejt genomja más-más variánst kapott. A HDR elősegítése érdekében a ligáz4 gént is kiütötték, ezzel kikapcsolva az NHEJ javítási útvonalat – ez a lépés Findlay szerint háromszoros hatékonyságnövekedést eredményezett. Végül, mivel minden sejt kopogtatása különböző, mélyen szekvenálták a sejteket, több milliószor lefedve ugyanazt a genomi régiót, hogy megbizonyosodjanak arról, hogy valóban azt a 4000 variánst kopogtatták be, amelyet tanulmányozni akartak. Két időpontban szekvenáltak, és arra következtettek, hogy azok a knock-inek, amelyek a második időpontban nem kerültek elő a szekvenálás során, olyanok voltak, amelyek beavatkoztak a gén működésébe, mert az őket hordozó sejteknek el kellett pusztulniuk.
Kipróbáld! Findlay csapata a 4000 variánshoz tartozó DNS-oligókat gyártatta le számukra egy microarray-n. Lehet 6000-250 000 oligót tartalmazó tömböket vásárolni, ezért Findlay szerint érdemes megfontolni, hogy több kísérletet kombinálva ugyanazon a tömbön több pénzt kapjunk a pénzünkért. Az ő laboratóriumuk körülbelül 5000 dollárt fizet 100 000 oligóért.
Ez a stratégia korlátozásokkal jár: eddig csak egynukleotid-variánsok kiütésére használták, és az összes módosításnak ugyanabban a génben kell lennie. A módszer akkor működik a legjobban, ha egy meglehetősen keskeny DNS-régiót, körülbelül 110-120 bázispárt szerkesztenek, mert a hosszabb DNS-oligók túl sok hibát tartalmaznának, mondja Findlay. Az is fontos, hogy nagyon mélyen szekvenáljunk, hogy biztosak lehessünk abban, hogy a beütni kívánt változatok teljes számát figyelembe vesszük.