ABOVE: modificat din © ISTOCK.com, tera vector
Aproape întotdeauna, a construi ceva este mai greu decât a-l dărâma. În mod similar, introducerea de gene reprezintă o provocare mai mare decât eliminarea lor. Este o realitate pe care cercetătorii vor trebui să o depășească pentru a obține cele mai bune rezultate din editarea genelor. Knocking in gene permite oamenilor de știință să studieze efectele unor variante genetice specifice, să utilizeze gene reporter, cum ar fi proteina verde fluorescentă, pentru a urmări produsele genice în timp și spațiu, să sondeze reglarea genomului și, în cele din urmă, să repare genele care cauzează boli. „Este o modalitate foarte eficientă de a interoga fiecare bază a unei gene”, spune Greg Findlay, doctorand în medicină/doctorand la Universitatea din Washington.
CRISPR-Cas9, o tehnologie de editare a genelor cunoscută pentru ușurința de utilizare, poate introduce sau elimina genele. Eliminarea unei gene presupune inserarea CRISPR-Cas9 într-o celulă cu ajutorul unui ARN ghid care direcționează instrumentul către gena de interes. Acolo, Cas9 taie gena, tăind ambele șiruri de ADN, iar mecanismul obișnuit de reparare a ADN-ului din celulă repară tăietura cu ajutorul unui proces numit NHEJ (non-homologous end joining). NHEJ este foarte eficient, dar imprecis. Procesul tinde să introducă erori sub forma unor mici inserții sau deleții care, de obicei, sunt suficiente pentru a elimina gena.
Pentru a elimina o genă, cu toate acestea, tăieturile trebuie să fie reparate foarte precis, fără inserții sau deleții suplimentare. Acest lucru necesită valorificarea unui al doilea mecanism de reparare a ADN-ului numit reparare dirijată de homologie (HDR), care – cel puțin în celulele mamiferelor – are loc mai puțin eficient, astfel încât frecvența sa este diminuată de cea a NHEJ. Complicând și mai mult procesul este faptul că unii loci genetici și anumite tipuri de celule sunt în mod inerent mai puțin ospitaliere pentru editarea CRISPR-Cas9.
În ultimii ani, cercetătorii au dezvoltat multe strategii noi pentru a spori eficiența operației de eliminare a genelor mari și mici cu ajutorul CRISPR-Cas9 și, pe parcurs, au propus și testat noi aplicații pentru acest tip de editare genetică. Aici, The Scientist explorează câteva dintre cele mai promițătoare abordări.
Select It
Cercetător: Jon Chesnut, director senior de biologie sintetică R&D, Thermo Fisher Scientific
Proiect: În dezvoltarea unui kit de marcare a genelor numit Truetag pe care Thermo Fisher îl va lansa pe piață la sfârșitul acestui an, Chesnut a folosit markeri selectibili pentru a îmbunătăți
eficiența. Un marker selectibil – în acest caz, o genă de rezistență la antibiotice – este lipit de un marker de proteină fluorescentă și introdus în
celule de mamifere. Aceste celule sunt apoi crescute în cultură cu antibioticul asociat. Gena de rezistență conferă un avantaj selectiv celulelor care o poartă; numai acestea sunt capabile să crească și, astfel, cele care cresc conțin marcajul genetic de interes. Chiar dacă eficiența inserției genei este scăzută, cercetătorii pot folosi selecția cu antibiotic timp de o săptămână sau mai mult pentru a ajunge la un procent ridicat de celule cu inserții reușite.
Utilizând antibioticul puromicină sau blasticidină cu kitul, echipa lui Chesnut a reușit să crească rata de inserție a genei de la 10-30% la 90% sau mai mult în unele populații de celule. Câteva gene deosebit de dificile au trecut de la o rată de inserție de mai puțin de 1 la sută la mai mult de 90 la sută. Este important să testați mai multe doze de antibiotice pe linia celulară pe care intenționați să o utilizați pentru a găsi doza corectă, spune Chesnut: doriți să ucideți celulele fără inserții, dar nu și celulele cu inserții reușite.
Încercați: Markerii selectibili funcționează cel mai bine atunci când gena de interes este puternic exprimată, spune Chesnut. „Dacă nu este, este posibil să obțineți în continuare selecție, dar este posibil să nu obțineți o expresie suficientă a markerului proteinei fluorescente pentru a o putea detecta.” De asemenea, se aplică limitările generale ale CRISPR-Cas9. „Există regiuni ale genomului care nu se taie foarte bine cu CRISPR și încă nu suntem siguri de ce”, adaugă el. Și unele tipuri de celule nu acceptă cu ușurință ADN străin, ARN sau complexe ARN-proteine – cele trei metode de livrare a CRISPR-Cas9.
Pentru a avea mai mult noroc în inserarea markerilor selectibili, asigurați-vă că există o așa-numită secvență PAM, o scurtă etichetă în ADN-ul țintă pe care CRISPR-Cas9 trebuie să o recunoască înainte de a tăia, la o distanță de 10 perechi de baze de locul de inserție a genei dorite, spune Chesnut. Mai departe de locul de tăiere decât atât, iar eficiența inserției poate fi prea mică pentru a fi funcțională. Fără un site PAM, puteți încerca TALEN-urile sau nucleazele cu degete de zinc, deși aceste tehnici mai vechi de editare a genelor sunt mai dificile decât CRISPR.
Timed Inhibition
Cercetător: Jacob Corn, biolog genomolog, Institutul Federal Elvețian de Tehnologie, Zurich
Proiect: Cercetătorii nu înțeleg de ce calea NHEJ surclasează în mare măsură calea HDR în celulele mamiferelor. „Drojdia face HDR ca o nebunie”, spune Corn. În efortul de a accelera acest proces de reparare a ADN-ului în celulele umane și de a îmbunătăți controlul genei knock-in, el și echipa sa încearcă să identifice modul în care este reglată HDR. Ei au analizat celulele umane pentru a găsi genele a căror knockdown a dus la o creștere a HDR în celulă și apoi au căutat inhibitori cu molecule mici ai acestor gene. Una dintre genele care au apărut codifică CDC7, o kinază care reglează tranziția ciclului celular la faza S; inhibitorul său, XL413, a sporit de două sau trei ori eficiența knock-in-ului genelor (BioRXiv, DOI: 10.1101/500462, 2018). Acest lucru se datorează faptului că HDR are loc doar în anumite părți ale ciclului celular, inclusiv în faza S, spune Corn. Dacă adăugați inhibitorul XL413 în același timp în care utilizați CRISPR-Cas9 pentru a edita gena țintă, celulele se îngrămădesc în faza imediat anterioară fazei S. Apoi eliminați XL413, iar toate celulele intră în faza S și crește eficiența knock-in.

Corn a folosit această tehnică în multe linii celulare umane imortalizate și în celulele T umane. Ea poate să facă knock in pe porțiuni scurte de ADN, cum ar fi SNP-urile, precum și gene mari. Nu există niciun motiv pentru care nu ar trebui să funcționeze la șoareci, spune el, deși nu a testat-o.
Încercați: „Sincronizarea este absolut esențială”, spune Corn. Cas9 trebuie să taie ADN-ul în același timp în care este adăugat XL413. Dacă inhibați mai întâi și apoi eliberați în timp ce editați cu CRISPR-Cas9, eficiența recombinării omoloage scade de trei ori în loc să crească, deoarece celulele sunt eliberate în faza greșită a ciclului celular.
Și, la fel ca în cazul oricărui efort HDR, spune Corn, efectuați întotdeauna un control fără niclază pentru a vă asigura că nu amplificați accidental ADN contaminant care plutește prin laborator. După introducerea knock-in-ului, „secvențiați, secvențiați, secvențiați, secvențiați”, spune el. Simpla utilizare a unui sistem reporter, cum ar fi un marker de proteină fluorescentă, pentru a demonstra inserția cu succes a genei poate fi contraproductivă. Secvențierea verifică dacă inserțiile au fost făcute la locul corect.
Playing the Long Game
Cercetător: Channabasavaiah Gurumurthy, director al instalației de bază de inginerie a genomului șoricelului, University of Nebraska Medical Center
Proiect: Cu câțiva ani în urmă, meditând asupra dificultății de a introduce gene în timp ce încerca să facă acest lucru în zigoții de șoarece, Gurumurthy și colegii săi au avut o revelație.
Cercetătorii reușeau să insereze cu succes ADN scurt, monocatenar, așa că de ce să nu încerce să facă un knock-in prin inserarea de ADN lung, monocatenar? Într-adevăr, abordarea, pe care Gurumurthy o numește Easi-CRISPR (adaosuri eficiente cu inserții de ADN ssDNA -CRISPR), sporește eficiența de 2,5 ori, iar utilizarea ADN monocatenar reduce de 100 de ori rata inserțiilor off-target în cultura celulară (Nat Protoc 13:195-215, 2018; Nature 559:405-09, 2018). „Este destul de uriaș”, spune el. În laboratorul lui Gurumurthy, Easi-CRISPR a generat o linie de șoareci knock-in pentru 9 din fiecare 10 gene pe care le-au încercat. Un colaborator a folosit-o, de asemenea, în celulele T umane pentru a crea celule CAR-T, celule imunitare specifice pacientului pentru combaterea cancerului.
Try It: Easi-CRISPR este departe de a fi infailibilă, avertizează Gurumurthy. Uneori, tehnica inserează doar o parte din genă. De asemenea, adaugă el, poate încurca brațele de omologie – secvențele scurte de o parte și de alta a genei care o orientează către ținta sa corectă în genom. Iar unii loci sunt în mod inexplicabil mai greu de inserat decât alții.
Puțini vânzători comerciali proiectează și sintetizează ADN personalizat lung, monocatenar. Vă puteți face propriul ADN, dar stabilitatea ADN-ului monocatenar variază; secvențele mai puțin stabile vor avea randamente mai mici, astfel încât este posibil să fie nevoie să sintetizați mai multe, spune Gurumurthy.
Cercetătorii care nu pot insera CRISPR în embrioni de șoarece unicelular pot plăti o instalație centrală pentru a face șoareci cu secvența lor de ADN, spune Gurumurthy. Facilitățile de bază, precum cea a lui, cer între 5.000 și 15.000 de dolari pentru a genera una sau două perechi de reproducere; facilitățile comerciale cer între 20.000 și 50.000 de dolari, spune el.
Knock-in By Numbers
Cercetător: Greg Findlay, doctorand în medicină/doctorand în laboratorul lui Jay Shendure, Universitatea din Washington
Proiect: Findlay și colegii săi urmăreau să îmbunătățească modul în care medicii interpretează mutațiile din gena BRCA1 a cancerului mamar și ovarian. Această genă are mii de variante, dar cercetătorii nu știu cum afectează majoritatea dintre ele funcția sa. Pentru a studia impactul acestor variante, ei au folosit o tehnică knock-in pe care au dezvoltat-o, numită editare genomică prin saturație (Nature, 562:217-22, 2018).
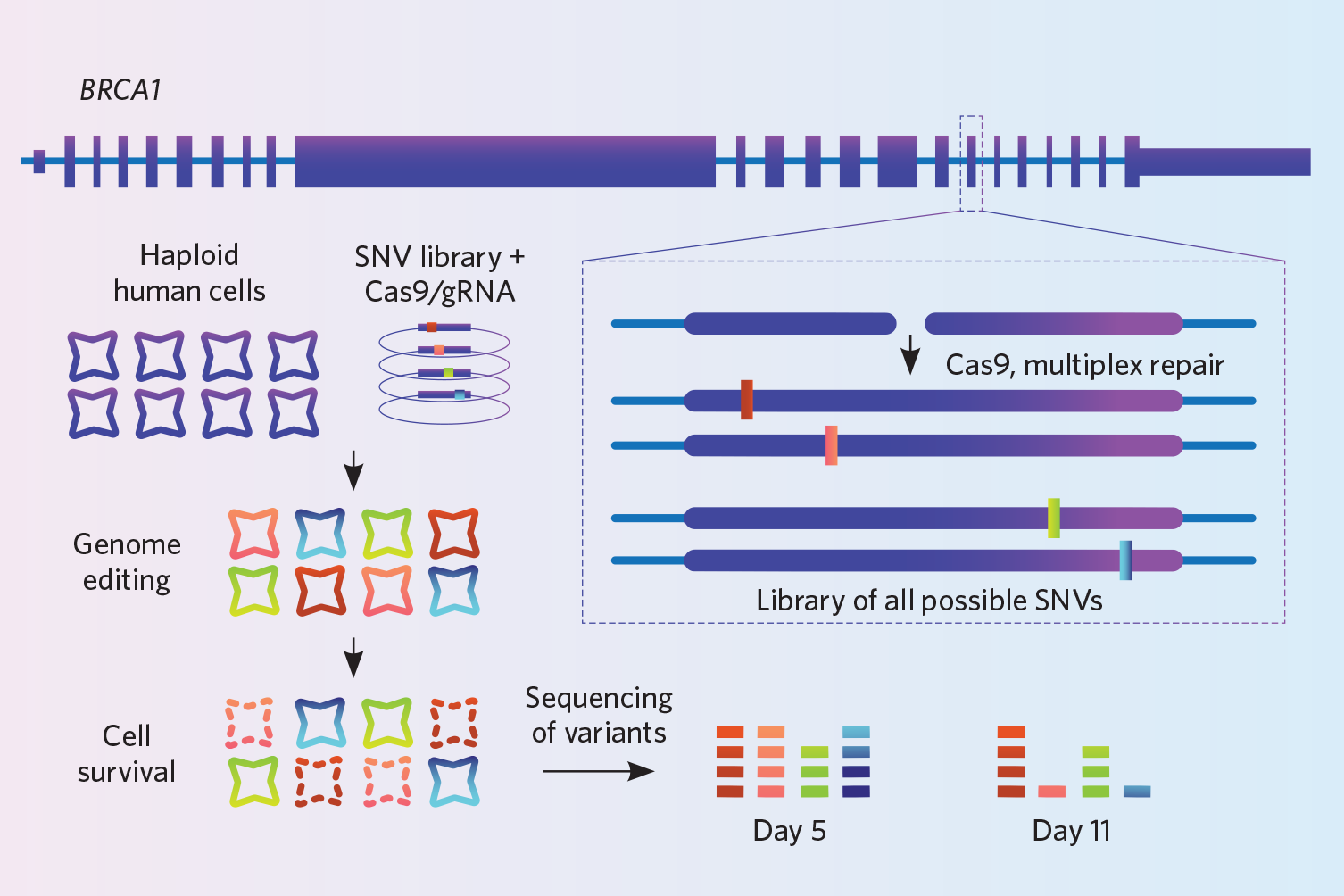
Într-o linie celulară umană haploidă imortalizată, ei au folosit CRISPR-Cas9 pentru a introduce 4.000 de mici variante în milioane de celule deodată in vitro. Genomul este tăiat în același loc în fiecare celulă, dar genomul fiecărei celule primește o variantă diferită. Pentru a promova HDR, ei au eliminat, de asemenea, gena ligase4, dezactivând calea de reparare NHEJ – un pas care a dus la o creștere de trei ori a eficienței, spune Findlay. În cele din urmă, deoarece toate knock-in-urile celulelor sunt diferite, au secvențiat celulele în profunzime, acoperind aceeași regiune genomică de milioane de ori, pentru a se asigura că au introdus efectiv cele 4.000 de variante pe care doreau să le studieze. Ei au secvențiat în două momente și au dedus că knock-in-urile care nu au apărut în secvențierea de la al doilea moment au fost cele care au interferat cu funcția genei, deoarece celulele care le purtau trebuie să fi murit.
Încercați: Echipa lui Findlay a avut oligoelemente de ADN pentru cele 4.000 de variante fabricate pentru ei pe un microarray. Puteți cumpăra matrici de 6.000 până la 250.000 de oligoelemente, așa că luați în considerare posibilitatea de a obține mai mult pentru banii dvs. prin combinarea mai multor experimente pe aceeași matrice, spune Findlay. Laboratorul lor plătește aproximativ 5.000 de dolari pentru 100.000 de oligo.
Această strategie vine cu limitări: până acum a fost folosită doar pentru a elimina variante cu un singur nucleotid, iar toate modificările trebuie să fie în aceeași genă. Metoda funcționează cel mai bine atunci când se editează o regiune destul de îngustă de ADN, aproximativ 110-120 de perechi de baze, deoarece oligoelementele de ADN mai lungi ar avea prea multe erori, spune Findlay. De asemenea, este important să secvențiați foarte profund pentru a vă asigura că luați în considerare întregul număr de variante pe care intenționați să le introduceți.
.