Nos últimos 10 anos, houve uma explosão de interesse em “computação científica” e “ciência de dados”: ou seja, a aplicação da computação para responder a perguntas e analisar dados nas ciências naturais e sociais. Para responder a estas necessidades, assistimos a um renascimento das linguagens de programação, ferramentas e técnicas que ajudam cientistas e investigadores a explorar e compreender dados e conceitos científicos, e a comunicar as suas descobertas. Mas até hoje, muito poucas ferramentas se concentraram em ajudar os cientistas a obter acesso não filtrado a todo o potencial de comunicação dos modernos navegadores da web. Portanto, hoje estamos entusiasmados em apresentar Iodide, uma ferramenta experimental destinada a ajudar os cientistas a escrever belos documentos interativos usando tecnologias web, tudo dentro de um fluxo de trabalho iterativo que se parece com outros ambientes de computação científica.

Iodide em ação.
Além de ser apenas um ambiente de programação para criar documentos vivos no navegador, Iodide tenta remover o atrito dos fluxos de trabalho comunicativos, sempre juntando a ferramenta de edição com o documento limpo e legível. Isto difere dos ambientes estilo IDE que produzem documentos de apresentação como arquivos .pdf (que são então divorciados do código original) e notebooks baseados em células que misturam código e elementos de apresentação. Em Iodide, você pode obter tanto um documento com a aparência que você quiser, quanto acesso fácil ao código subjacente e ao ambiente de edição.
Iodide ainda está muito em estado alfa, mas seguindo o aforismo da internet “Se você não está envergonhado com a primeira versão do seu produto, você lançou tarde demais”, decidimos fazer um lançamento suave muito cedo, na esperança de obter feedback de uma comunidade maior. Temos uma demo que você pode experimentar agora mesmo, mas espere um monte de arestas vivas (e por favor não use este lançamento alfa para trabalhos críticos!). Estamos esperando que, apesar das asperezas, se você se esgueire para isso você será capaz de ver o valor do conceito, e que o feedback que você nos dá nos ajudará a descobrir onde ir em seguida.
- Como chegamos a Iodide
- Data science at Mozilla
- Por que há tão pouca rede na ciência?
- Towards Iodide
- A anatomia de Iodide
- As Visões de Exploração e Relatório
- Vivo, documentos interativos com o poder da Plataforma Web
- Partilha, colaboração e reprodutibilidade
- Pyodide: A pilha de ciência Python no browser
- JSMD (JavaScript MarkDown)
- O que vem a seguir?
- Características avançadas de colaboração
- Mais idiomas!
- Exportar arquivo de notebook
- Iodide to text editor browser extension
- Feedback e colaboração bem-vindos!
- Sobre Brendan Colloran
Como chegamos a Iodide
Data science at Mozilla
No Mozilla, a grande maioria do nosso trabalho de ciência de dados é focado na comunicação. Apesar de algumas vezes implantarmos modelos destinados a melhorar diretamente a experiência do usuário, como o mecanismo de recomendação que ajuda os usuários a descobrir extensões de navegador, na maioria das vezes nossos cientistas de dados analisam nossos dados a fim de encontrar e compartilhar insights que irão informar as decisões dos gerentes de produto, engenheiros e executivos.
Data science work involves writing a lot of code, but unlike traditional software development, our objective is to answer questions, not to produce software. Isso normalmente resulta em algum tipo de relatório – um documento, alguns gráficos, ou talvez uma visualização de dados interativa. Como muitas organizações de ciência de dados, na Mozilla nós exploramos nossos dados usando ferramentas fantásticas como Jupyter e R-Studio. Entretanto, quando é hora de compartilhar nossos resultados, normalmente não podemos entregar um caderno Jupyter ou um script R para um tomador de decisão, então frequentemente acabamos fazendo coisas como copiar índices e estatísticas resumidas para um Google Doc.
Descobrimos que fazer a viagem de ida e volta desde explorar dados em código até criar uma explicação digerível e voltar novamente nem sempre é fácil. Pesquisas mostram que muitas pessoas compartilham essa experiência. Quando um cientista de dados está lendo o relatório final de outro e quer olhar o código por trás dele, pode haver muita fricção; às vezes rastrear o código é fácil, às vezes não. Se eles querem tentar experimentar e estender o código, as coisas obviamente ficam mais difíceis ainda. Outro cientista de dados pode ter seu código, mas pode não ter uma configuração idêntica em sua máquina, e a configuração leva tempo.
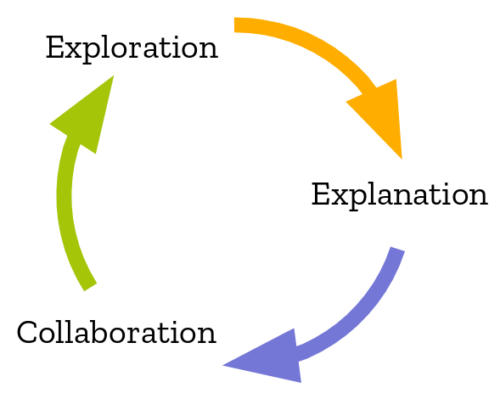
O círculo virtuoso do trabalho de ciência de dados.
Por que há tão pouca rede na ciência?
Against the background of these data science workflows at Mozilla, no final de 2017 eu empreendi um projeto que exigia a visualização interativa de dados. Hoje você pode criar visualizações interativas usando grandes bibliotecas para Python, R, e Julia, mas para o que eu queria realizar, eu precisava cair no Javascript. Isto significava afastar-me dos meus ambientes favoritos de ciência de dados. As ferramentas de desenvolvimento web modernas são incrivelmente poderosas, mas extremamente complicadas. Eu realmente não queria descobrir como obter uma cadeia de ferramentas de construção Javascript completa, com o módulo quente recarregando e rodando, mas sem isso eu não consegui encontrar muito voltado para a criação de documentos web limpos e legíveis dentro do fluxo de trabalho ao vivo e iterativo que me é familiar.
Comecei a me perguntar por que essa ferramenta não existia – por que não há Jupyter para a construção de documentos web interativos – e logo fiz zoom out para pensar por que quase ninguém usa Javascript para computação científica. Três grandes razões saltaram:
- Javascript em si tem uma reputação mista entre os cientistas por ser lento e desajeitado;
- não há muitas bibliotecas de computação científica que rodam no navegador ou que funcionam com Javascript; e,
- como descobri, há muito poucas ferramentas de codificação científica que permitem um loop de iteração rápido e também dão acesso não filtrado às capacidades de apresentação no navegador.
Estes são desafios muito grandes. Mas como pensei mais sobre isso, comecei a pensar que trabalhar em um navegador pode ter algumas vantagens reais para o tipo de ciência de dados comunicativos que fazemos no Mozilla. A maior vantagem, claro, é que o browser tem indiscutivelmente o conjunto mais avançado e bem suportado de tecnologias de apresentação do planeta, do DOM ao WebGL, do Canvas ao WebVR.
Pensando na fricção do fluxo de trabalho mencionada acima, outra potencial vantagem me ocorreu: no browser, o documento final não precisa ser separado da ferramenta que o criou. Eu queria uma ferramenta projetada para ajudar os cientistas a iterar em documentos web (basicamente aplicativos web de um único propósito para explicar uma idéia)… e muitas ferramentas que estávamos usando eram basicamente aplicativos web. Para o caso de uso da escrita desses pequenos documentos web, porque não juntar o documento com a ferramenta usada para escrevê-lo?
Ao fazer isso, leitores não técnicos podiam ver meu documento bonito, mas outros cientistas de dados podiam voltar instantaneamente ao código original. Além disso, como o kernel de computação seria o motor JS do navegador, eles seriam capazes de começar a estender e experimentar o código de análise imediatamente. E eles seriam capazes de fazer tudo isso sem se conectar a recursos de computação remota ou instalar qualquer software.
Towards Iodide
Comecei a discutir os potenciais prós e contras da computação científica no navegador com meus colegas, e no decorrer de nossas conversas, notamos algumas outras tendências interessantes.
No Mozilla estávamos vendo muitas demos interessantes mostrando o WebAssembly, uma nova maneira dos navegadores executarem código escrito em outros idiomas além do Javascript. WebAssembly permite que os programas sejam executados a uma velocidade incrível, em alguns casos próxima aos binários nativos. Nós estávamos vendo exemplos de processos computacionalmente caros como motores de jogo 3D inteiros rodando dentro do navegador sem dificuldade. Seguindo em frente, seria possível compilar as melhores bibliotecas de computação numérica C e C++ para o WebAssembly e envolvê-las em APIs JS ergonômicas, assim como o projeto SciPy faz para o Python. De fato, projetos já tinham começado a fazer isso.
WebAssembly torna possível executar código em velocidade quase nativa no navegador.
Também notamos a vontade da comunidade Javascript de introduzir nova sintaxe ao fazer isso ajuda as pessoas a resolverem seus problemas de forma mais eficaz. Talvez fosse possível emular alguns dos elementos sintáticos chave que tornam a programação numérica mais compreensível e fluida no MATLAB, Julia e Python – multiplicação de matrizes, corte multidimensional, operações de broadcast array, e assim por diante. Mais uma vez, encontramos outras pessoas pensando de forma semelhante.
Com estas linhas convergindo, começamos a nos perguntar se a plataforma web poderia estar à beira de se tornar uma casa produtiva para a computação científica. No mínimo, parecia que poderia evoluir para servir alguns dos fluxos de trabalho comunicativos que encontramos no Mozilla (e que tantos outros encontram na indústria e na academia). Com o núcleo do Javascript a melhorar constantemente e a possibilidade de adicionar extensões de sintaxe para a programação numérica, talvez o próprio JS pudesse ser mais apelativo para os cientistas. A WebAssembly parecia oferecer um caminho para grandes bibliotecas científicas. A terceira parte do banco de dados seria um ambiente para a criação de documentos científicos de dados para a web. Este último elemento é onde nós decidimos focar nossa experimentação inicial, que nos levou a Iodide.
A anatomia de Iodide
Iodide é uma ferramenta projetada para dar aos cientistas um fluxo de trabalho familiar para a criação de documentos interativos de grande aparência usando todo o poder da plataforma web. Para conseguir isso, damos-lhe um “relatório” – basicamente uma página web que pode preencher com o seu conteúdo – e algumas ferramentas para explorar dados iterativamente e modificar o seu relatório para criar algo que esteja pronto a partilhar. Quando estiver pronto, você pode enviar um link diretamente para o seu relatório finalizado. Se os seus colegas e colaboradores quiserem rever o seu código e aprender com ele, podem voltar para um modo de exploração com um clique. Se eles quiserem experimentar o código e usá-lo como base do seu próprio trabalho, com mais um clique eles podem forká-lo e começar a trabalhar na sua própria versão.
Ler um pouco mais sobre algumas das ideias que estamos a experimentar numa tentativa de tornar este fluxo de trabalho fluido.
As Visões de Exploração e Relatório
Iodeto pretende apertar o loop entre exploração, explicação e colaboração. O central para isso é a habilidade de se mover para frente e para trás entre uma boa aparência de escrita e um ambiente útil para exploração computacional iterativa.
Quando você cria um novo notebook Iodide pela primeira vez, você começa na “vista de exploração”. Isto fornece um conjunto de painéis incluindo um editor para escrever código, um console para visualizar a saída do código que você avalia, um visualizador de espaço de trabalho para examinar as variáveis que você criou durante a sua sessão, e um painel de “visualização de relatório” no qual você pode ver uma visualização do seu relatório.

Editando um trecho de código Markdown no view de exploração de Iodide.
Ao clicar no botão “REPORT” no canto superior direito, o conteúdo da pré-visualização do seu relatório irá expandir-se para preencher toda a janela, permitindo que você coloque a história que você quer contar na frente e no centro. Os leitores que não sabem como codificar ou que não estão interessados nos detalhes técnicos são capazes de se concentrar no que você está tentando transmitir sem ter que percorrer o código. Quando um leitor visita o link para a visualização do relatório, o seu código será executado automaticamente. se quiser rever o seu código, basta clicar no botão “EXPLORAR” no canto superior direito para trazê-los de volta para a visualização de exploração. A partir daí, eles podem fazer uma cópia do notebook para suas próprias explorações.

Movendo da vista explorar para a vista de relatório.
Quando você compartilha um link para um notebook Iodide, seu colaborador pode sempre acessar essas duas vistas. O documento limpo e legível nunca é separado do código executável subjacente e do ambiente de edição ao vivo.
Vivo, documentos interativos com o poder da Plataforma Web
Documentos Iodide ao vivo no navegador, o que significa que o mecanismo de computação está sempre disponível. Sempre que você compartilha seu trabalho, você compartilha um relatório interativo ao vivo com o código em execução. Além disso, como o cálculo acontece no navegador junto com a apresentação, não há necessidade de chamar um backend de idioma em outro processo. Isto significa que documentos interativos são atualizados em tempo real, abrindo a possibilidade de visualizações 3D sem interrupção, mesmo com a baixa latência e alta taxa de quadros necessária para VR.

Contribuidor Devin Bayly explora dados de RM do seu cérebro
Partilha, colaboração e reprodutibilidade
Building Iodide na web simplifica uma série de elementos de fricção do fluxo de trabalho que encontramos em outras ferramentas. A partilha é simplificada porque a escrita e o código estão disponíveis na mesma URL em vez de, digamos, colar um link para um script nas notas de rodapé de um Google Doc. A colaboração é simplificada porque o kernel de computação é o navegador e as bibliotecas podem ser carregadas através de um pedido HTTP como qualquer página da web carrega script – não é necessário instalar idiomas, bibliotecas ou ferramentas adicionais. E como os navegadores fornecem uma camada de compatibilidade, você não precisa se preocupar com o comportamento do notebook sendo reproduzível entre computadores e SOs.
Para suportar fluxos de trabalho colaborativos, construímos um servidor bastante simples para salvar e compartilhar notebooks. Há uma instância pública no iodide.io onde você pode experimentar com o Iodide e compartilhar seu trabalho publicamente. Também é possível configurar sua própria instância atrás de um firewall (e de fato é isso que já estamos fazendo no Mozilla para algum trabalho interno). Mas o importante é que os próprios notebooks não estão profundamente ligados a uma única instância do servidor Iodide. Se a necessidade surgir, deve ser fácil migrar seu trabalho para outro servidor ou exportar seu notebook como um pacote para compartilhar em outros serviços como Netlify ou Github Pages (mais sobre a exportação de pacotes abaixo em “O que vem a seguir?”). Manter a computação no cliente nos permite focar na construção de um ambiente realmente ótimo para compartilhamento e colaboração, sem a necessidade de construir recursos computacionais na nuvem.
Pyodide: A pilha de ciência Python no browser
Quando começamos a pensar em tornar a web melhor para os cientistas, focamos em maneiras que poderíamos melhorar o trabalho com Javascript, como compilar bibliotecas científicas existentes para WebAssembly e envolvê-las em APIs JS fáceis de usar. Quando propusemos isso aos feiticeiros do Mozilla WebAssembly, eles ofereceram uma idéia mais ambiciosa: se muitos cientistas preferem Python, encontre-os onde eles estão compilando a pilha científica Python para rodar em WebAssembly.
Pensamos que isso soou assustador – que seria um projeto enorme e que nunca entregaria um desempenho satisfatório… mas duas semanas depois Mike Droettboom teve uma implementação funcional do Python rodando dentro de um notebook Iodide. Nos próximos dois meses, adicionamos Numpy, Pandas e Matplotlib, que são de longe os módulos mais utilizados no ecossistema científico Python. Com a ajuda dos colaboradores Kirill Smelkov e Roman Yurchak na Nexedi, conseguimos suporte para Scipy e Scikit-learn. Desde então, nós continuamos adicionando outras bibliotecas pouco a pouco.
Executar o interpretador Python dentro de uma máquina virtual Javascript adiciona uma penalidade de performance, mas essa penalidade acaba sendo surpreendentemente pequena – em nossos benchmarks, cerca de 1x-12x mais lento que o nativo no Firefox e 1x-16x mais lento no Chrome. A experiência mostra que isso é muito útil para a exploração interativa.

Executar o Matplotlib no navegador permite suas características interativas, que não estão disponíveis em ambientes estáticos
Bringing Python no navegador cria alguns fluxos de trabalho mágicos. Por exemplo, você pode importar e limpar seus dados em Python, e então acessar os objetos Python resultantes a partir do Javascript (na maioria dos casos, a conversão acontece automaticamente) para que você possa exibi-los usando bibliotecas JS como d3. Ainda mais magicamente, você pode acessar as APIs do navegador a partir do código Python, permitindo que você faça coisas como manipular o DOM sem tocar no Javascript.
Obviamente, há muito mais a dizer sobre Pyodide, e ele merece um artigo próprio – vamos entrar em mais detalhes em um post de acompanhamento no próximo mês.
JSMD (JavaScript MarkDown)
Apenas como no modo Jupyter e R-Markdown do R, em Iodide você pode intercalar código e escrever como quiser, quebrando seu código em “pedaços de código” que você pode modificar e executar como unidades separadas. Nossa implementação desta idéia é paralela ao R Markdown e ao “modo celular” do MATLAB: ao invés de usar uma interface explicitamente baseada em células, o conteúdo de um notebook Iodide é apenas um documento de texto que usa uma sintaxe especial para delimitar tipos específicos de células. Chamamos este formato de texto de “JSMD”.
MATLAB, os pedaços de código são definidos por linhas começando com %% seguidas por uma string indicando a linguagem do pedaço abaixo. Atualmente suportamos trechos contendo Javascript, CSS, Markdown (e HTML), Python, um trecho especial “fetch” que simplifica o carregamento de recursos, e um trecho de plugin que permite estender a funcionalidade do Iodide adicionando novos tipos de células.
Achamos este formato bastante conveniente. Ele torna fácil o uso de ferramentas orientadas a texto como diff viewers e seu próprio editor de texto favorito, e você pode realizar operações de texto padrão como cortar/copiar/colar sem ter que aprender atalhos para o gerenciamento de células. Para mais detalhes você pode ler sobre JSMD em nosso docs.
O que vem a seguir?
Vale a pena repetir que ainda estamos em alfa, então continuaremos a melhorar o polimento geral e a eliminar bugs. Mas, além disso, temos uma série de características em mente para a nossa próxima ronda de experiências. Se alguma destas ideias saltar como particularmente útil, avise-nos! Melhor ainda, deixe-nos saber se você gostaria de nos ajudar a construí-las!
Características avançadas de colaboração
Como mencionado acima, até agora construímos um backend muito simples que lhe permite salvar seu trabalho online, olhar para o trabalho feito por outras pessoas, e rapidamente garfo e extensão de notebooks existentes feitos por outros usuários, mas estes são apenas os passos iniciais de um útil fluxo de trabalho colaborativo.
Os próximos três grandes recursos de colaboração que estamos analisando são:
- Google Docs-selementos de comentários
- A capacidade de sugerir alterações ao notebook de outro usuário através de um fluxo de trabalho de fork/merge similar ao Github pull requests
- Edição simultânea de notebooks como o Google Docs.
Neste momento, estamos priorizando-os mais ou menos nessa ordem, mas se você os resolveria numa ordem diferente ou se tiver outras sugestões, nos avise!
Mais idiomas!
Falamos com pessoas das comunidades R e Julia sobre a compilação desses idiomas para o WebAssembly, o que permitiria seu uso em Iodide e outros projetos baseados em navegadores. Nossa investigação inicial indica que isso deveria ser possível, mas que implementar essas linguagens pode ser um pouco mais desafiador do que Python. Como no Python, alguns fluxos de trabalho legais se abrem se você puder, por exemplo, encaixar modelos estatísticos em R ou resolver equações diferenciais em Julia, e então exibir seus resultados usando as APIs do navegador. Se trazer essas linguagens para a web lhe interessa, por favor, procure – em particular, nós adoraríamos a ajuda dos experts em FORTRAN e LLVM.
Exportar arquivo de notebook
Versões iniciais do Iodide eram arquivos HTML auto-contidos, que incluíam tanto o código JSMD usado na análise, quanto o código JS usado para executar o próprio Iodide, mas nós nos afastamos dessa arquitetura. Experiências posteriores nos convenceram de que os benefícios de colaboração de ter um servidor Iodide superam as vantagens de gerenciar arquivos em seu sistema local. No entanto, estes experimentos nos mostraram que é possível tirar uma foto executável de um notebook Iodide, inserindo o código Iodide juntamente com quaisquer dados e bibliotecas usadas por um notebook em um grande arquivo HTML. Isto pode acabar sendo um arquivo maior do que você gostaria de servir para usuários regulares, mas pode ser útil como um snapshot perfeitamente reproduzível e arquivável de uma análise.
Iodide to text editor browser extension
Embora muitos cientistas estejam bastante acostumados a trabalhar em ambientes de programação baseados em browser, nós sabemos que algumas pessoas nunca irão editar código em nada além do seu editor de texto favorito. Nós realmente queremos que Iodide conheça pessoas onde elas já estão, incluindo aquelas que preferem digitar seu código em outro editor, mas querem acesso aos recursos interativos e iterativos que Iodide fornece. Para atender a essa necessidade, começamos a pensar em criar uma extensão de navegador leve e algumas APIs simples para deixar Iodide falar com os editores do lado do cliente.
Feedback e colaboração bem-vindos!
Não estamos tentando resolver todos os problemas da ciência de dados e computação científica, e sabemos que Iodide não será a xícara de chá de todos. Se você precisa processar terabytes de dados em clusters de GPU, Iodide provavelmente não tem muito a oferecer a você. Se você está publicando artigos de jornal e só precisa escrever um documento LaTeX, então existem ferramentas melhores para as suas necessidades. Se toda a tendência de trazer coisas para o navegador faz você se encolher um pouco, não há problema – há uma série de ferramentas realmente incríveis que você pode usar para fazer ciência, e nós estamos gratos por isso! Não queremos mudar a maneira como ninguém trabalha, e para muitos cientistas a comunicação focada na web está fora de questão. Rad! Viva sua melhor vida!
Mas para aqueles cientistas que produzem conteúdo para a web, e para aqueles que gostariam de fazê-lo se você tivesse ferramentas projetadas para suportar a forma como você trabalha: nós realmente adoraríamos ouvir de você!
Por favor, visite iodide.io, experimente e nos dê feedback (mas novamente: tenha em mente que este projeto está em fase alfa – por favor, não o use para nenhum trabalho crítico, e por favor, esteja ciente de que enquanto estamos em fase alfa tudo está sujeito a mudanças). Você pode fazer nossa pesquisa rápida, e os problemas do Github e relatórios de bugs são muito bem-vindos. Pedidos de recursos e pensamentos sobre a direção geral podem ser compartilhados através do nosso grupo Google ou Gitter.
Se você gostaria de se envolver em nos ajudar a construir Iodide, nós somos de código aberto no Github. Iodide toca uma grande variedade de disciplinas de software, desde o desenvolvimento de frontend moderno até a computação científica, compilação e transposição, por isso há muitas coisas interessantes a fazer! Por favor, procure se alguma destas coisas lhe interessa!
Muito obrigado a Hamilton Ulmer, William Lachance e Mike Droettboom pelo seu grande trabalho sobre Iodide e por rever este artigo.
Sobre Brendan Colloran
Outros artigos de Brendan Colloran…