ABOVE: aangepast van © ISTOCK.com, tera vector
Nagenoeg altijd is iets opbouwen moeilijker dan iets afbreken. Zo is het ook een grotere uitdaging om genen in te bouwen dan uit te schakelen. Het is een realiteit die onderzoekers zullen moeten overwinnen om het meeste te halen uit het bewerken van genen. Het inkloppen van genen stelt wetenschappers in staat om de effecten van specifieke genvarianten te bestuderen, om reportergenen zoals groen fluorescent eiwit te gebruiken om genproducten in tijd en ruimte te volgen, om genoomregulering te onderzoeken, en uiteindelijk, om ziekteveroorzakende genen te herstellen. “Het is echt een effectieve manier om elke base van een gen te ondervragen,” zegt Greg Findlay, een MD/PhD kandidaat aan de Universiteit van Washington.
CRISPR-Cas9, een genbewerkingstechnologie die bekend staat om zijn gebruiksvriendelijkheid, kan genen in- of uitschakelen. Het uitschakelen van een gen houdt in dat CRISPR-Cas9 in een cel wordt ingebracht met behulp van een gids-RNA dat het gereedschap richt op het gen van interesse. Cas9 knipt het gen door beide DNA-strengen heen en het normale DNA-reparatiemechanisme van de cel herstelt de knip met behulp van een proces dat non-homologous end joining (NHEJ) wordt genoemd. NHEJ is zeer efficiënt maar onnauwkeurig. Het proces heeft de neiging om fouten te introduceren in de vorm van kleine inserties of deleties die gewoonlijk voldoende zijn om het gen uit te schakelen.
Om een gen in te schakelen, moeten de sneden echter zeer nauwkeurig worden gerepareerd, zonder extra inserties of deleties. Dit vereist de inzet van een tweede DNA-herstelmechanisme, homologie-gestuurd herstel (HDR), dat – althans in zoogdiercellen – minder efficiënt voorkomt, zodat de frequentie ervan in het niet valt bij die van NHEJ. Het proces wordt verder bemoeilijkt door het feit dat sommige genloci en celtypes inherent minder geschikt zijn voor CRISPR-Cas9 editing.
In de afgelopen jaren hebben onderzoekers veel nieuwe strategieën ontwikkeld om de efficiëntie van het inkloppen van zowel grote als kleine genen met CRISPR-Cas9 te verhogen, en gaandeweg hebben ze nieuwe toepassingen voor dit type genbewerking voorgesteld en getest. Hier onderzoekt The Scientist een paar van de meest veelbelovende benaderingen.
Select It
Onderzoeker: Jon Chesnut, senior director of synthetic biology R&D, Thermo Fisher Scientific
Project: Bij de ontwikkeling van een “gene tagging kit”, Truetag genaamd, die Thermo Fisher later dit jaar op de markt zal brengen, gebruikte Chesnut selecteerbare merkers om de
efficiëntie te verbeteren. Een selecteerbare merker – in dit geval een antibiotica-resistentiegen – wordt aan een fluorescente proteïnetag geplakt en in zoogdiercellen geklopt. Deze cellen worden vervolgens gekweekt in een cultuur met het bijbehorende antibioticum. Het resistentiegen geeft een selectief voordeel aan de cellen die het dragen; alleen zij zijn in staat te groeien, en dus bevatten de cellen die groeien de genmarkering van belang. Zelfs als de efficiëntie van de geninsertie laag is, kunnen onderzoekers antibioticaselectie gedurende een week of langer gebruiken om te eindigen met een hoog percentage cellen met succesvolle inserties.
Door het antibioticum puromycine of blasticidin met de kit te gebruiken, slaagde Chesnut’s team erin het geninsertiepercentage op te voeren van 10-30 procent tot 90 procent of meer in sommige celpopulaties. Een paar bijzonder moeilijke genen gingen van een invoegingspercentage van minder dan 1 procent naar meer dan 90 procent. Het is belangrijk om meerdere doses antibiotica te testen op de cellijn die je van plan bent te gebruiken om de juiste dosis te vinden, zegt Chesnut: je wilt cellen zonder inserties doden, maar geen cellen met succesvolle inserties.
Probeer het: Selecteerbare markers werken het beste wanneer het gen van interesse sterk tot expressie komt, zegt Chesnut. “Als dat niet het geval is, kun je nog steeds selectie krijgen, maar misschien krijg je niet genoeg expressie van je fluorescerende eiwit tag om het te kunnen detecteren.” Ook zijn de algemene beperkingen van CRISPR-Cas9 van toepassing. “Er zijn gebieden van het genoom die niet goed snijden met CRISPR, en we weten nog steeds niet zeker waarom,” voegt hij eraan toe. En sommige celtypes accepteren niet gemakkelijk vreemd DNA, RNA, of RNA-eiwit complexen – de drie methoden van CRISPR-Cas9 levering.
Voor meer geluk met het invoegen van selecteerbare markers, zorg ervoor dat er een zogenaamde PAM-sequentie is, een korte tag in het doel-DNA dat CRISPR-Cas9 moet herkennen voordat het snijdt, binnen 10 baseparen van de gewenste gen invoegplaats, zegt Chesnut. Verder weg van de snijplaats kan de invoegingsefficiëntie te laag zijn om functioneel te zijn. Zonder een PAM-locatie kun je TALENs of zinkvingernucleasen proberen, hoewel die oudere genbewerkingstechnieken lastiger zijn dan CRISPR.
Timed Inhibition
Onderzoeker: Jacob Corn, genoombioloog, Zwitsers Federaal Instituut voor Technologie, Zürich
Project: Onderzoekers begrijpen niet waarom het NHEJ-pad het HDR-pad in zoogdiercellen ver achter zich laat. “Gisten doen HDR als een gek,” zegt Corn. In een poging om dit DNA-herstelproces in menselijke cellen op te voeren en de controle op gen knock-in te verbeteren, proberen hij en zijn team vast te stellen hoe HDR wordt gereguleerd. Ze onderzochten menselijke cellen op genen waarvan de uitschakeling leidde tot een verhoogde HDR in de cel, en zochten vervolgens naar kleine molecule-remmers van die genen. Een van de genen die naar voren kwam codeert voor CDC7, een kinase dat de overgang van de celcyclus naar de S-fase regelt; de remmer ervan, XL413, verhoogde de efficiëntie van gen knock-in twee- tot drievoudig (BioRXiv, DOI: 10.1101/500462, 2018). Dat komt omdat HDR slechts in enkele delen van de celcyclus voorkomt, waaronder de S-fase, zegt Corn. Als je de remmer XL413 toevoegt op hetzelfde moment dat je CRISPR-Cas9 gebruikt om je doelgen te bewerken, stapelen de cellen zich op in de fase vlak voor de S-fase. Als je XL413 vervolgens verwijdert, gaan alle cellen naar de S-fase en neemt de knock-in-efficiëntie toe.

Corn heeft deze techniek gebruikt in veel geïmmortaliseerde menselijke cellijnen en in menselijke T-cellen. Korte stukken DNA, zoals SNP’s, maar ook grote genen kunnen er mee worden geklopt. Er is geen reden waarom het niet zou werken bij muizen, zegt hij, hoewel hij het nog niet heeft getest.
Probeer het: “Timing is absoluut de sleutel,” zegt Corn. Cas9 moet het DNA afsnijden op hetzelfde moment dat XL413 wordt toegevoegd. Als je eerst inhibeert en dan vrijgeeft tijdens het bewerken met CRISPR-Cas9, daalt de homologe recombinatie-efficiëntie drievoudig in plaats van toe te nemen, omdat de cellen in de verkeerde fase van de celcyclus worden vrijgegeven.
En zoals bij elke HDR-inspanning, zegt Corn, voer altijd een no-nuclease-controle uit om ervoor te zorgen dat je niet per ongeluk verontreinigend DNA amplificeert dat in je lab rondzweeft. Na het inbrengen van de knock-in, “sequentie, sequentie, sequentie, sequentie”, zegt hij. Het gebruik van een reporter zoals een fluorescerend eiwit om aan te tonen dat het gen succesvol is ingebracht, kan averechts werken. Sequencing verifieert dat de inserties op de juiste plaats hebben plaatsgevonden.
Playing the Long Game
Onderzoeker: Channabasavaiah Gurumurthy, directeur van de kernfaciliteit voor muizengenoomtechniek, University of Nebraska Medical Center
Project: Een paar jaar geleden, mijmerend over de moeilijkheid van het inkloppen van genen terwijl ze dat probeerden te doen in muis zygotes, hadden Gurumurthy en zijn collega’s een openbaring.
Onderzoekers slaagden erin kort, enkelstrengs DNA in te brengen, dus waarom niet proberen een knock-in te maken door lang, enkelstrengs DNA in te brengen? Inderdaad, de aanpak, die Gurumurthy Easi-CRISPR noemt (efficiënte toevoegingen met ssDNA-inserts -CRISPR), verhoogt de efficiëntie met 2,5 keer, en het gebruik van enkelstrengs DNA verlaagt de snelheid van off-target inserties 100-voudig in celkweek (Nat Protoc 13:195-215, 2018; Nature 559:405-09, 2018). “Het is behoorlijk enorm”, zegt hij. In het lab van Gurumurthy heeft Easi-CRISPR een knock-in muizenlijn gegenereerd voor 9 van elke 10 genen die ze hebben geprobeerd. Een medewerker heeft het ook gebruikt in menselijke T-cellen om CAR-T-cellen te maken, patiëntspecifieke immuuncellen voor het bestrijden van kanker.
Probeer het: Easi-CRISPR is verre van onfeilbaar, waarschuwt Gurumurthy. Soms voegt de techniek slechts een deel van het gen in. Ook, voegt hij eraan toe, kan het de homologie armen – de korte sequenties aan weerszijden van het gen dat het huis aan zijn juiste doel in het genoom. En sommige loci zijn onverklaarbaar moeilijker in te voegen dan andere.
Weinig commerciële verkopers ontwerpen en synthetiseren op maat gemaakt lang, enkelstrengs DNA. Je kunt je eigen maken, maar de stabiliteit van enkelstrengs DNA varieert; minder stabiele sequenties zullen lagere opbrengsten hebben, dus je moet er misschien meer van synthetiseren, zegt Gurumurthy.
Onderzoekers die niet in staat zijn om CRISPR in te brengen in eencellige muizenembryo’s kunnen een kernfaciliteit betalen om de muizen te maken met hun DNA-sequentie, zegt Gurumurthy. Kernfaciliteiten zoals de zijne rekenen $5.000-$15.000 voor het genereren van een of twee fokparen; commerciële faciliteiten rekenen $20.000-$50.000, zegt hij.
Knock-in by Numbers
Onderzoeker: Greg Findlay, MD/PhD kandidaat in het lab van Jay Shendure, Universiteit van Washington
Project: Findlay en zijn collega’s wilden de manier verbeteren waarop clinici mutaties in het borst- en eierstokkankergen BRCA1 interpreteren. Dat gen heeft duizenden varianten, maar onderzoekers weten niet hoe de meeste daarvan de functie ervan beïnvloeden. Om de impact van deze varianten te bestuderen, gebruikten ze een knock-in techniek die ze ontwikkelden genaamd saturation genome editing (Nature, 562:217-22, 2018).
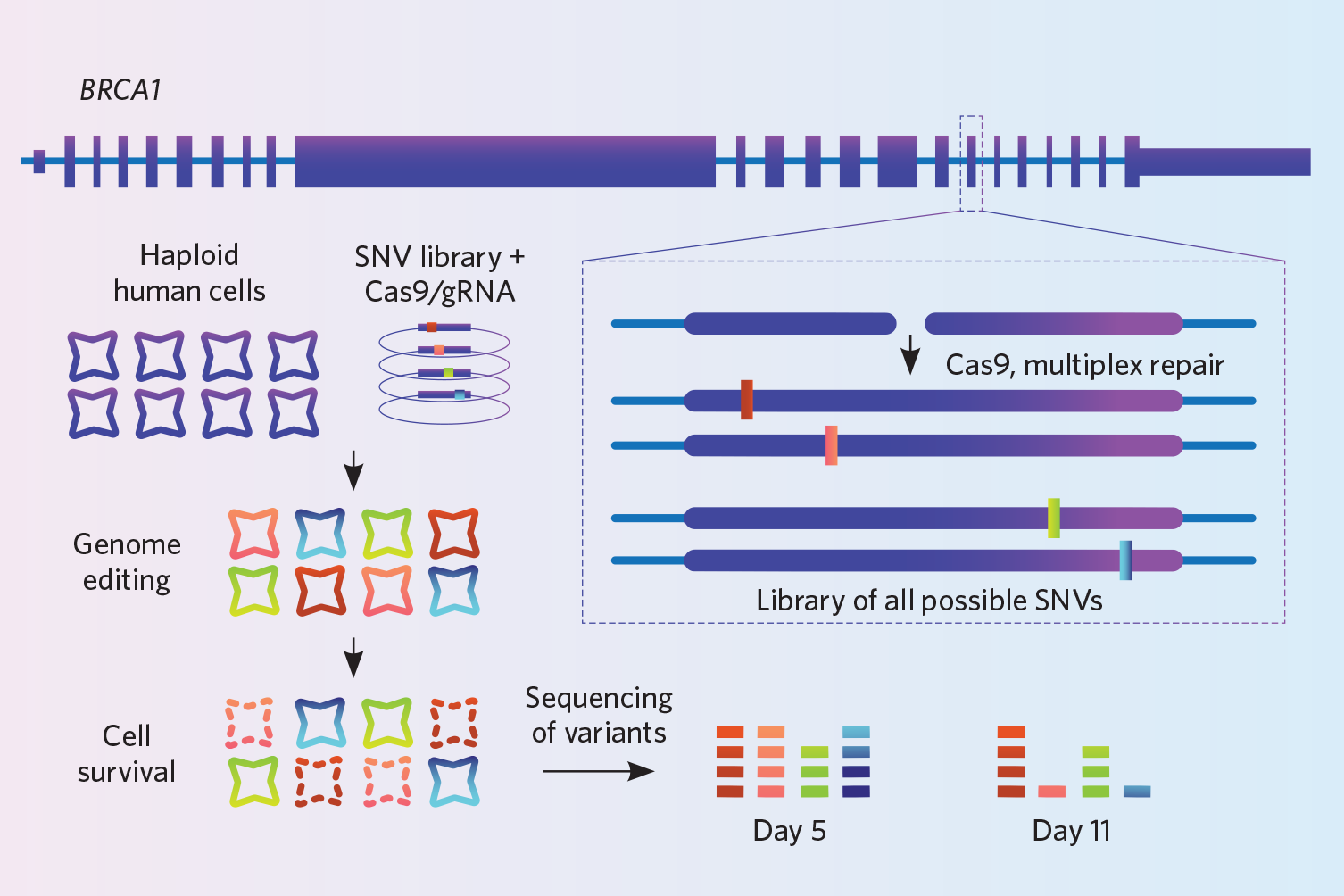
In een geïmmortaliseerde haploïde menselijke cellijn gebruikten ze CRISPR-Cas9 om in vitro 4.000 kleine varianten in miljoenen cellen tegelijk in te kloppen. Het genoom wordt in elke cel op dezelfde plaats doorgesneden, maar elke cel krijgt een andere variant. Om HDR te bevorderen, schakelden ze ook het ligase4-gen uit, waardoor het NHEJ-herstelpad werd uitgeschakeld – een stap die volgens Findlay een drievoudige efficiëntiewinst opleverde. Aangezien alle knock-ins in de cellen verschillend zijn, hebben ze de cellen grondig gesequenced, waarbij ze dezelfde genomische regio miljoenen keren hebben bekeken, om er zeker van te zijn dat ze de 4.000 varianten die ze wilden bestuderen ook echt hadden ingebouwd. Ze namen de sequentie op twee tijdstippen en leidden daaruit af dat de knock-ins die op het tweede tijdstip niet in de sequentie naar voren kwamen, degenen waren die de functie van het gen verstoorden, omdat de cellen die deze varianten droegen, moeten zijn gestorven.
Probeer het: Findlay’s team heeft de DNA oligo’s voor de 4.000 varianten op een microarray laten maken. Je kunt arrays kopen van 6.000 tot 250.000 oligos, dus overweeg om meer waar voor je geld te krijgen door meerdere experimenten op dezelfde array te combineren, zegt Findlay. Hun lab betaalt ongeveer 5.000 dollar voor 100.000 oligos.
Deze strategie komt met beperkingen: het is tot nu toe alleen gebruikt om single-nucleotide varianten te kloppen, en alle bewerkingen moeten in hetzelfde gen zitten. De methode werkt het beste bij het bewerken van een vrij smal gebied van DNA, ongeveer 110-120 basenparen, omdat langere DNA-oligo’s te veel fouten zouden bevatten, aldus Findlay. Het is ook belangrijk om zeer diep te sequencen om er zeker van te zijn dat je rekening houdt met het volledige aantal varianten dat je wilde inkloppen.