Die stickstoffhaltigen Basen, wichtige Bestandteile der Nukleotide, sind organische Moleküle und werden so genannt, weil sie Kohlenstoff und Stickstoff enthalten. Sie sind Basen, weil sie eine Aminogruppe enthalten, die die Möglichkeit hat, einen zusätzlichen Wasserstoff zu binden und so die Wasserstoffionenkonzentration in ihrer Umgebung zu verringern, wodurch sie basischer werden. Jedes Nukleotid in der DNA enthält eine von vier möglichen stickstoffhaltigen Basen: Adenin (A), Guanin (G), Cytosin (C) und Thymin (T). RNA-Nukleotide enthalten ebenfalls eine der vier möglichen Basen: Adenin, Guanin, Cytosin und Uracil (U) anstelle von Thymin.
Adenin und Guanin werden als Purine klassifiziert. Die Primärstruktur eines Purins besteht aus zwei Kohlenstoff-Stickstoff-Ringen. Cytosin, Thymin und Uracil werden als Pyrimidine klassifiziert, die einen einzigen Kohlenstoff-Stickstoff-Ring als Primärstruktur haben (Abbildung 1). An jeden dieser grundlegenden Kohlenstoff-Stickstoff-Ringe sind verschiedene funktionelle Gruppen gebunden. In der Molekularbiologie werden die stickstoffhaltigen Basen einfach mit den Symbolen A, T, G, C und U bezeichnet. Die DNS enthält A, T, G und C, während die RNS A, U, G und C enthält.
Der Pentosezucker in der DNS ist die Desoxyribose und in der RNS ist der Zucker die Ribose (Abbildung 1). Der Unterschied zwischen den Zuckern ist das Vorhandensein einer Hydroxylgruppe am zweiten Kohlenstoff der Ribose und von Wasserstoff am zweiten Kohlenstoff der Desoxyribose. Die Kohlenstoffatome des Zuckermoleküls sind mit 1′, 2′, 3′, 4′ und 5′ nummeriert (1′ wird als „eine Primzahl“ gelesen). Der Phosphatrest ist an die Hydroxylgruppe des 5′-Kohlenstoffs eines Zuckers und an die Hydroxylgruppe des 3′-Kohlenstoffs des Zuckers des nächsten Nukleotids gebunden, wodurch eine 5′-3′-Phosphodiester-Bindung entsteht. Die Phosphodiester-Bindung wird nicht durch eine einfache Dehydratisierungsreaktion gebildet wie die anderen Bindungen, die Monomere in Makromolekülen verbinden: Ihre Bildung erfordert die Entfernung von zwei Phosphatgruppen. Ein Polynukleotid kann Tausende solcher Phosphodiesterbindungen aufweisen.
DNA-Doppelhelixstruktur
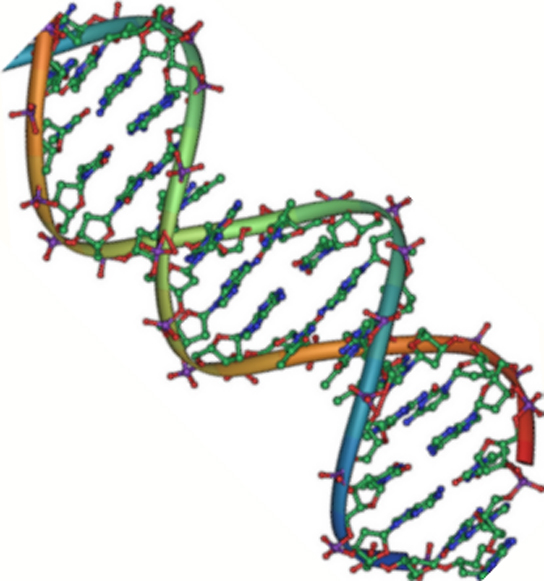
Abbildung 2. Die DNA ist eine antiparallele Doppelhelix. Das Phosphatgerüst (die geschwungenen Linien) befindet sich auf der Außenseite und die Basen sind auf der Innenseite. Jede Base interagiert mit einer Base des gegenüberliegenden Strangs. (credit: Jerome Walker/Dennis Myts)
Die DNA hat eine Doppelhelixstruktur (Abbildung 2). Der Zucker und das Phosphat liegen an der Außenseite der Helix und bilden das Rückgrat der DNA. Die stickstoffhaltigen Basen sind im Inneren wie die Stufen einer Treppe paarweise aufeinander gestapelt; die Paare sind durch Wasserstoffbrückenbindungen miteinander verbunden. Jedes Basenpaar in der Doppelhelix ist vom nächsten Basenpaar durch 0,34 nm getrennt.
Die beiden Stränge der Helix verlaufen in entgegengesetzter Richtung, was bedeutet, dass das 5′-Kohlenstoffende eines Stranges dem 3′-Kohlenstoffende seines Gegenstranges gegenüberliegt. (Dies wird als antiparallele Ausrichtung bezeichnet und ist wichtig für die DNA-Replikation und für viele Nukleinsäure-Interaktionen.)
Nur bestimmte Arten von Basenpaarungen sind erlaubt. Zum Beispiel kann sich ein bestimmtes Purin nur mit einem bestimmten Pyrimidin paaren. Das bedeutet, dass sich A mit T und G mit C paaren kann, wie in Abbildung 3 dargestellt. Dies ist als Basenkomplementärregel bekannt. Mit anderen Worten: Die DNA-Stränge sind komplementär zueinander. Wenn die Sequenz des einen Strangs AATTGGCC ist, hat der komplementäre Strang die Sequenz TTAACCGG. Während der DNA-Replikation wird jeder Strang kopiert, so dass eine Tochter-DNA-Doppelhelix entsteht, die einen elterlichen DNA-Strang und einen neu synthetisierten Strang enthält.
Praxisfrage
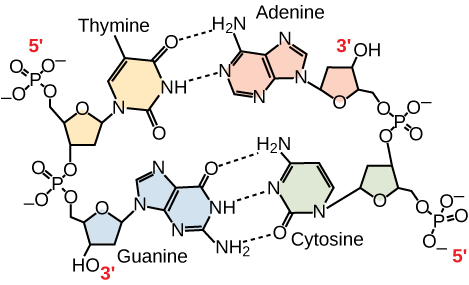
Abbildung 3. In einem doppelsträngigen DNA-Molekül verlaufen die beiden Stränge antiparallel zueinander, so dass ein Strang 5′ zu 3′ und der andere 3′ zu 5′ verläuft. Das Phosphatgerüst befindet sich an der Außenseite, die Basen liegen in der Mitte. Adenin bildet Wasserstoffbrücken (oder Basenpaare) mit Thymin, und Guanin bildet Basenpaare mit Cytosin.
Eine Mutation tritt auf, und Cytosin wird durch Adenin ersetzt. Welche Auswirkungen wird dies Ihrer Meinung nach auf die DNA-Struktur haben?
RNA
Ribonukleinsäure, oder RNA, ist hauptsächlich am Prozess der Proteinsynthese unter der Leitung der DNA beteiligt. RNA ist in der Regel einzelsträngig und besteht aus Ribonukleotiden, die durch Phosphodiesterbindungen verbunden sind. Ein Ribonukleotid in der RNA-Kette enthält Ribose (den Pentosezucker), eine der vier Stickstoffbasen (A, U, G und C) und die Phosphatgruppe.
Es gibt vier Haupttypen von RNA: Boten-RNA (mRNA), ribosomale RNA (rRNA), Transfer-RNA (tRNA) und microRNA (miRNA). Die erste, die mRNA, überträgt die Botschaft der DNA, die alle zellulären Aktivitäten in einer Zelle steuert. Wenn eine Zelle die Synthese eines bestimmten Proteins benötigt, wird das Gen für dieses Produkt „eingeschaltet“ und die Boten-RNA im Zellkern synthetisiert. Die Basensequenz der RNA ist komplementär zu der kodierenden Sequenz der DNA, von der sie kopiert wurde. Allerdings fehlt in der RNA die Base T und stattdessen ist U vorhanden. Wenn der DNA-Strang die Sequenz AATTGCGC hat, ist die Sequenz der komplementären RNA UUAACGCG. Im Zytoplasma interagiert die mRNA mit Ribosomen und anderen zellulären Maschinen (Abbildung 4).
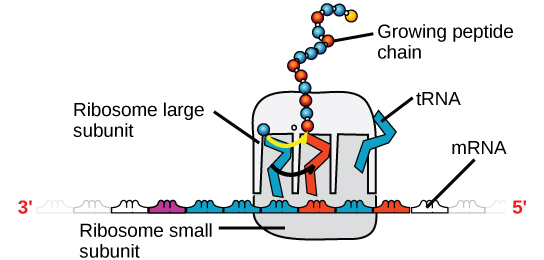
Abbildung 4. Ein Ribosom besteht aus zwei Teilen: einer großen Untereinheit und einer kleinen Untereinheit. Die mRNA befindet sich zwischen den beiden Untereinheiten. Ein tRNA-Molekül erkennt ein Codon auf der mRNA, bindet daran durch komplementäre Basenpaarung und fügt die richtige Aminosäure an die wachsende Peptidkette an.
Die mRNA wird in Gruppen von drei Basen gelesen, die als Codons bezeichnet werden. Jedes Codon kodiert für eine einzelne Aminosäure. Auf diese Weise wird die mRNA abgelesen und das Proteinprodukt hergestellt. Die ribosomale RNA (rRNA) ist ein Hauptbestandteil der Ribosomen, an die sich die mRNA bindet. Die rRNA sorgt für die richtige Ausrichtung der mRNA und der Ribosomen; die rRNA des Ribosoms hat auch eine enzymatische Aktivität (Peptidyltransferase) und katalysiert die Bildung der Peptidbindungen zwischen zwei ausgerichteten Aminosäuren. Die Transfer-RNA (tRNA) ist eine der kleinsten der vier RNA-Typen, in der Regel 70-90 Nukleotide lang. Sie transportiert die richtige Aminosäure zum Ort der Proteinsynthese. Es ist die Basenpaarung zwischen der tRNA und der mRNA, die es ermöglicht, die richtige Aminosäure in die Polypeptidkette einzufügen. microRNAs sind die kleinsten RNA-Moleküle und ihre Rolle besteht in der Regulierung der Genexpression, indem sie die Expression bestimmter mRNA-Botschaften stören.